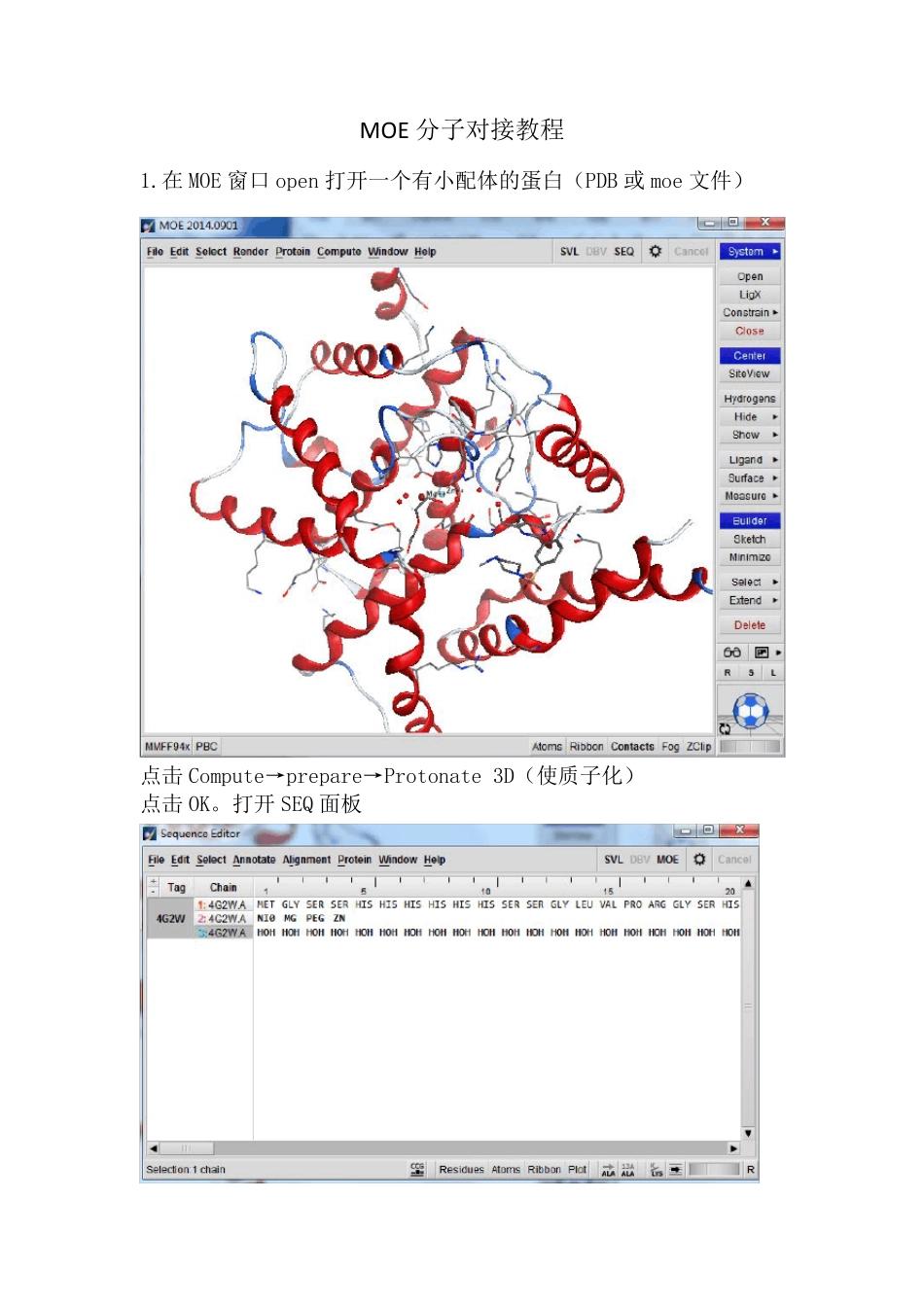
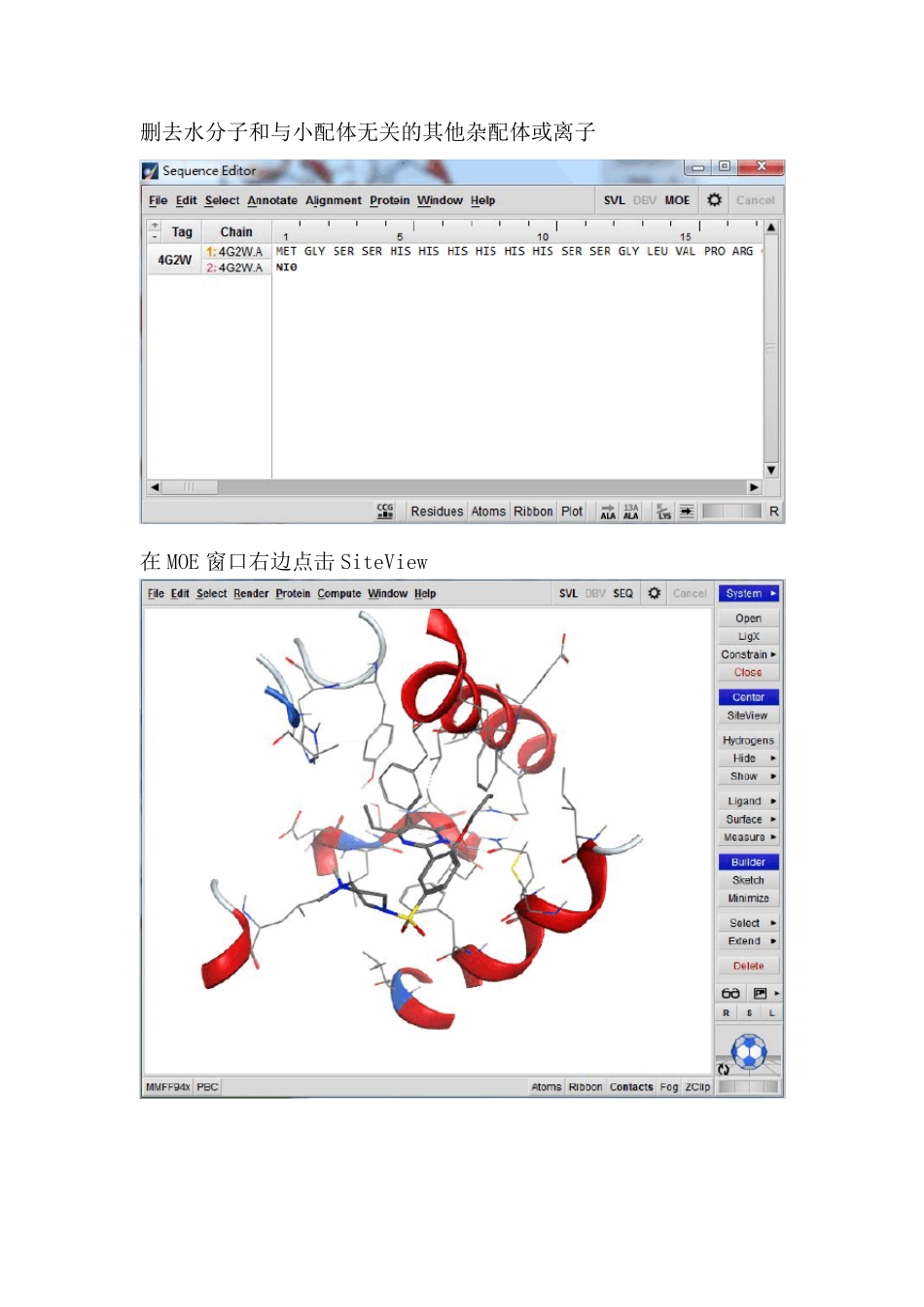
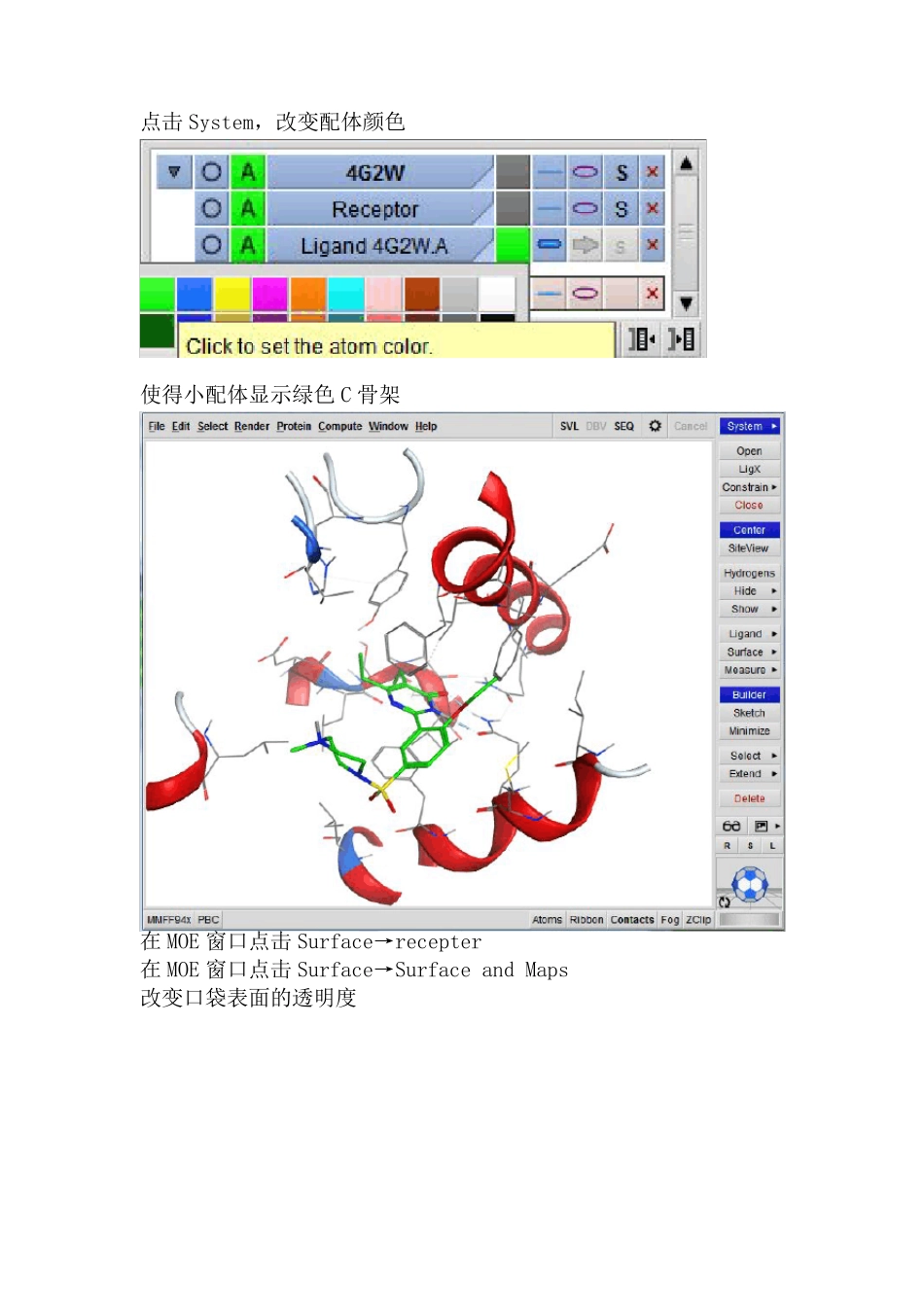
MOE 分子对接教程1.在MOE 窗口open 打开一个有小配体的蛋白(PDB 或moe 文件)点击Compute→prepare→Protonate 3D(使质子化)点击OK。打开SEQ 面板删去水分子和与小配体无关的其他杂配体或离子在 MOE 窗口右边点击 SiteView点击System,改变配体颜色使得小配体显示绿色C 骨架在MOE 窗口点击Surface→recepter在MOE 窗口点击Surface→Surface and Maps改变口袋表面的透明度在Surface and Maps 面板调节如图所示在MOE 窗口会看到如下图所示:接下来对接一个小配体点击Compute→dock在 Recepter 选项选择 Recepter+Solvent其它选择默认值。点击Run。在表单你会看到有不同的构象对接结果,包括 RMSD 值。口袋对接结果2.创建一个药效团模型在MOE 窗口点击Compute→Pharmacophore→Query Editor点击R,这样会创建基于受体的药效团特征。点击口袋中显示的小球,然后点击feature,这样在对接口袋就会显示药效团。3.化合物数据库的对接关闭Pharmacophore Query Editor 面板在MOE 窗口点击Compute→DockOutput 选项选择一个对接结果数据库名字,这里命名为 moe_dock2Receptor 选项为 Recepter+SolventLigand 选项为 MDB File,选择要对接的pde5inhibitors.mdb(这里说明一下,mdb 格式是 MOE 独有的,用的不多,但是可以将常用的sdf 和 mol2 格式转化为 mdb 格式,方法为将 sdf 或 mol2 文件用MOE 打开成表单,再 save as 成 mdb 格式。)其他值默认,点击Run。等待计算...对接完后,关闭药效团编辑器面板