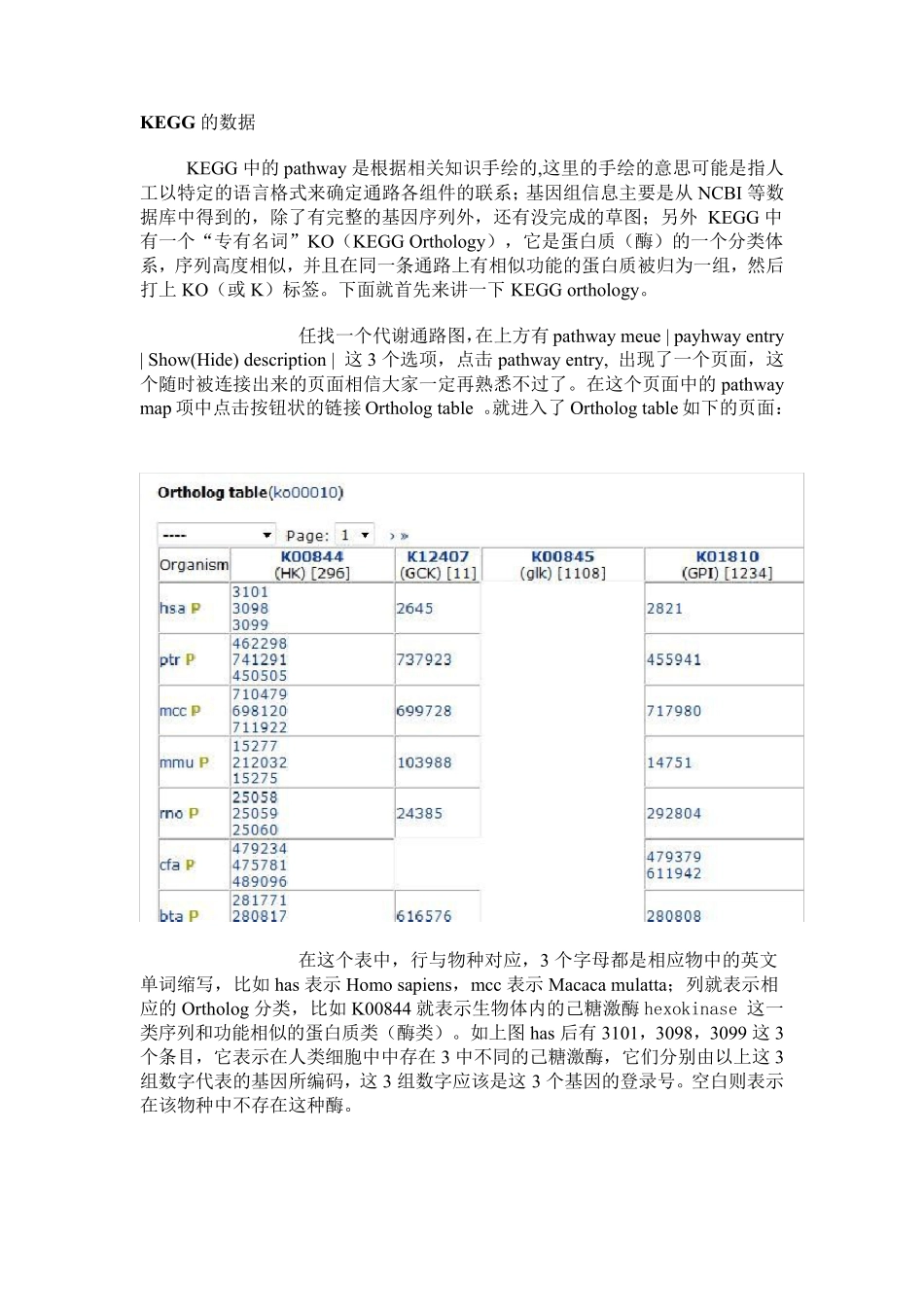
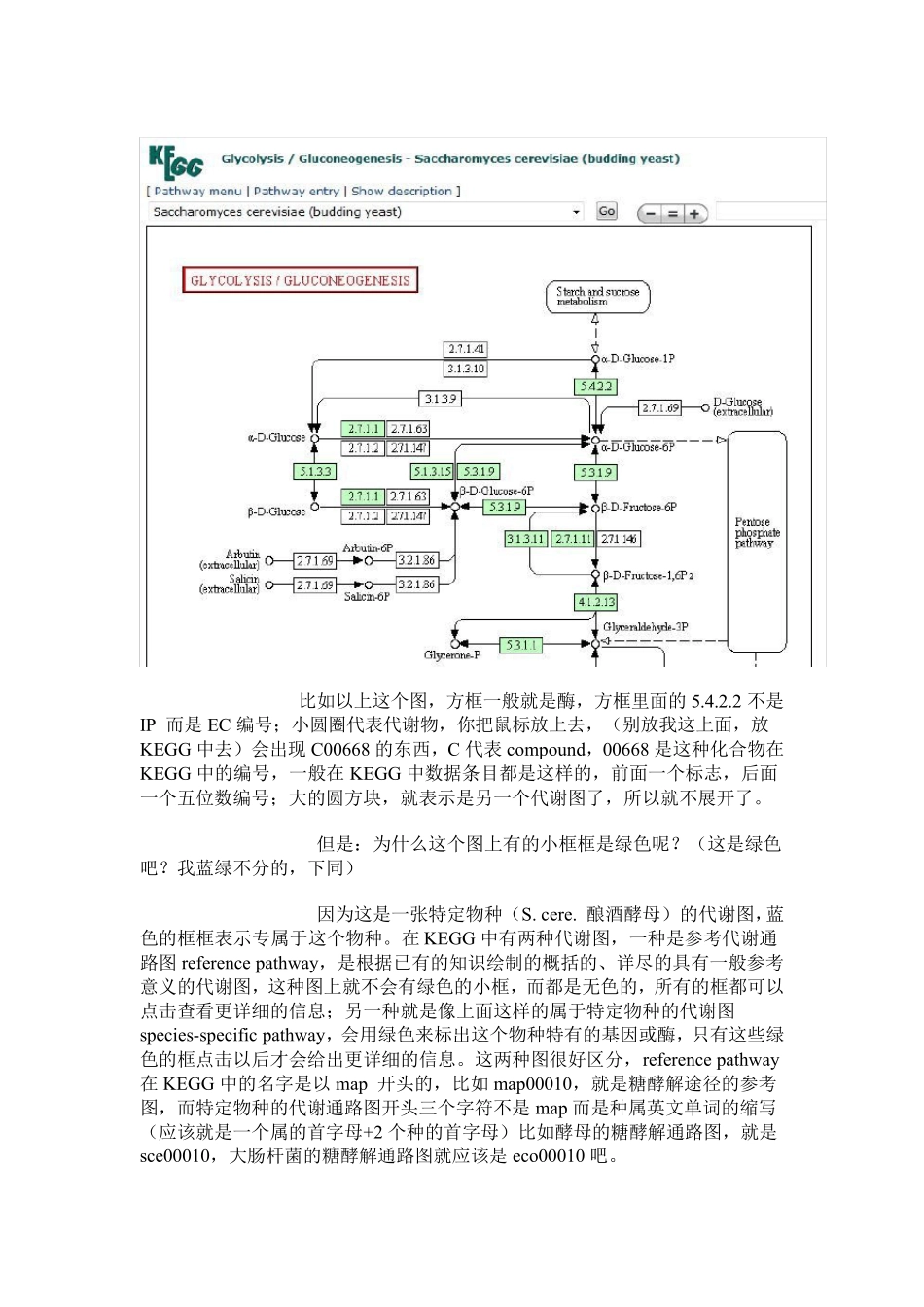
KEGG 的数据KEGG 中的pathway 是根据相关知识手绘的,这里的手绘的意思可能是指人工以特定的语言格式来确定通路各组件的联系;基因组信息主要是从NCBI 等数据库中得到的,除了有完整的基因序列外,还有没完成的草图;另外KEGG 中有一个“专有名词”KO(KEGG Orthology),它是蛋白质(酶)的一个分类体系,序列高度相似,并且在同一条通路上有相似功能的蛋白质被归为一组,然后打上 KO(或 K)标签。下面就首先来讲一下 KEGG orthology。任找一个代谢通路图,在上方有pathway meue |payhway entry|Show(Hide) description | 这3 个选项,点击 pathway entry, 出现了一个页面,这个随时被连接出来的页面相信大家一定再熟悉不过了。在这个页面中的pathwaymap 项中点击按钮状的链接 Ortholog table 。就进入了Ortholog table 如下的页面:在这个表中,行与物种对应,3 个字母都是相应物中的英文单词缩写,比如 has 表示 Homo sapiens,mcc 表示 Macaca mulatta;列就表示相应的Ortholog 分类,比如 K00844 就表示生物体内的己糖激酶 hexokinase 这一类序列和功能相似的蛋白质类(酶类)。如上图has 后有3101,3098,3099 这3个条目,它表示在人类细胞中中存在 3 中不同的己糖激酶,它们分别由以上这3组数字代表的基因所编码,这3 组数字应该是这3 个基因的登录号。空白则表示在该物种中不存在这种酶。点击K00844 则这一KO 分类信息及成员列表都可显示出来;点击has则链接到物种(人类)基因组去了;点击P,则显示相应的代谢通路。下面我们点击3101,如下:如上图,就是我们常见的一个页面,3101 是KEGG 中的基因ID(登录号),H.sapiens 表示物种,然后是基因的名称,表达的酶,属于哪个KO 分类以及参与哪些代谢途径;下面还有结构、序列信息等等。所以从 Ortholog table 中可以很容易地知道一张代谢通路上有哪些KO 分类(酶类),并且这些酶类的成员在各物种中分配存在的情况以及特定的名称。怎么看 KEGG 中代谢通路图比如以上这个图,方框一般就是酶,方框里面的5.4.2.2 不是IP 而是EC 编号;小圆圈代表代谢物,你把鼠标放上去,(别放我这上面,放KEGG 中去)会出现C00668 的东西,C 代表compound,00668 是这种化合物在KEGG 中的编号,一般在KEGG 中数据条目都是这样的,前面一个标志,后面一个五位数编号;大的圆方块,就表示是另一个代谢图了,所以就不展开了。但是:为什么...