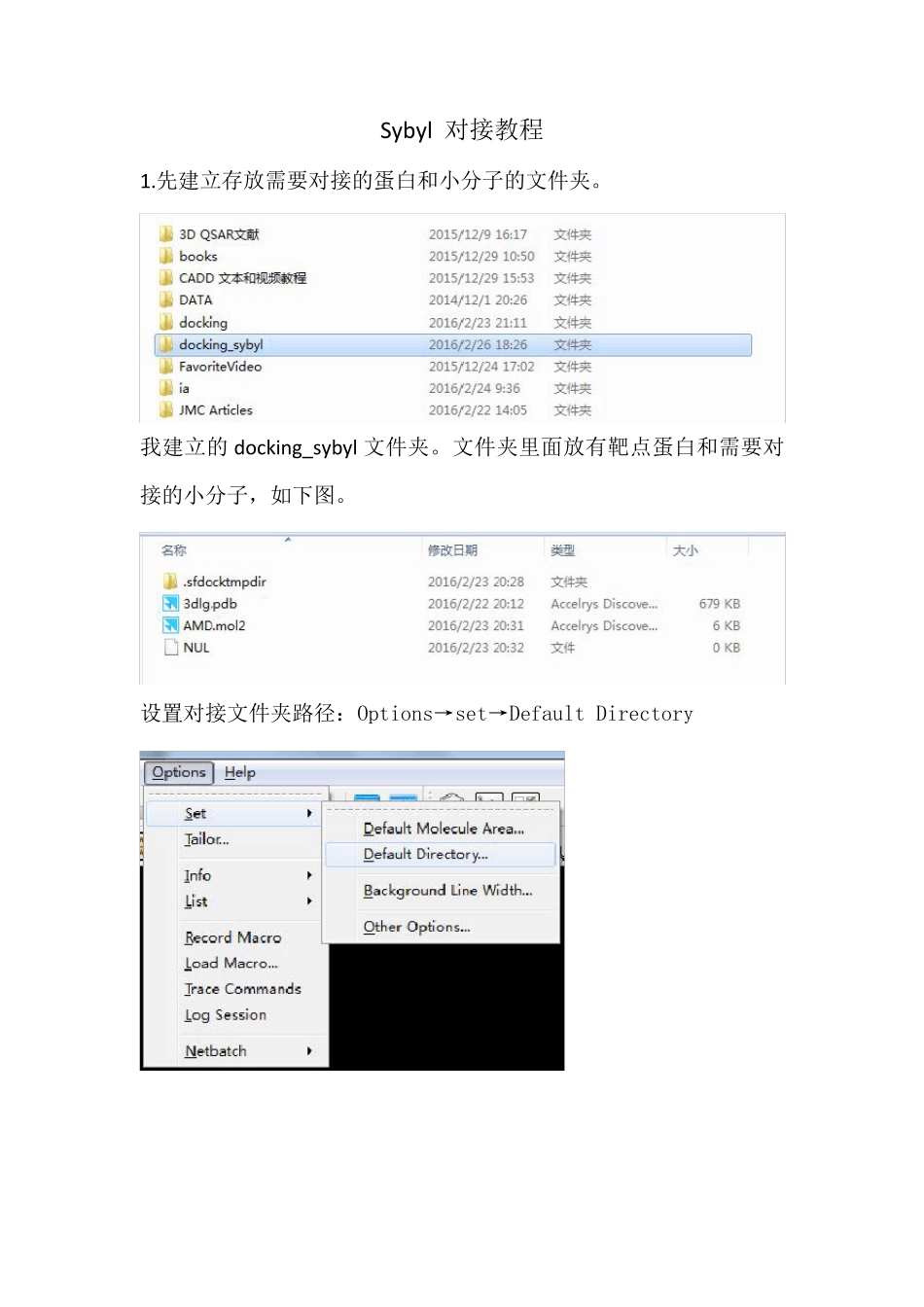
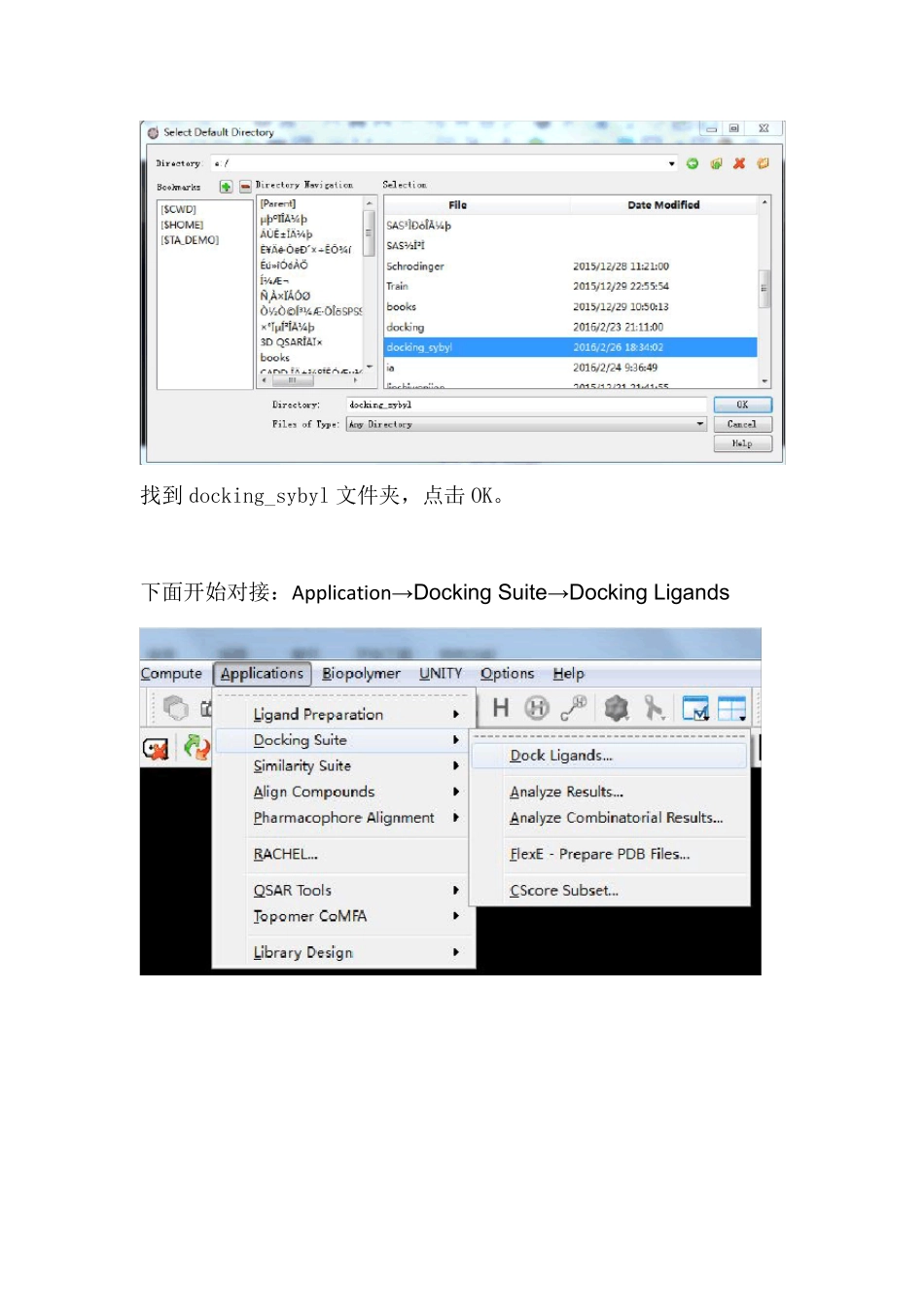
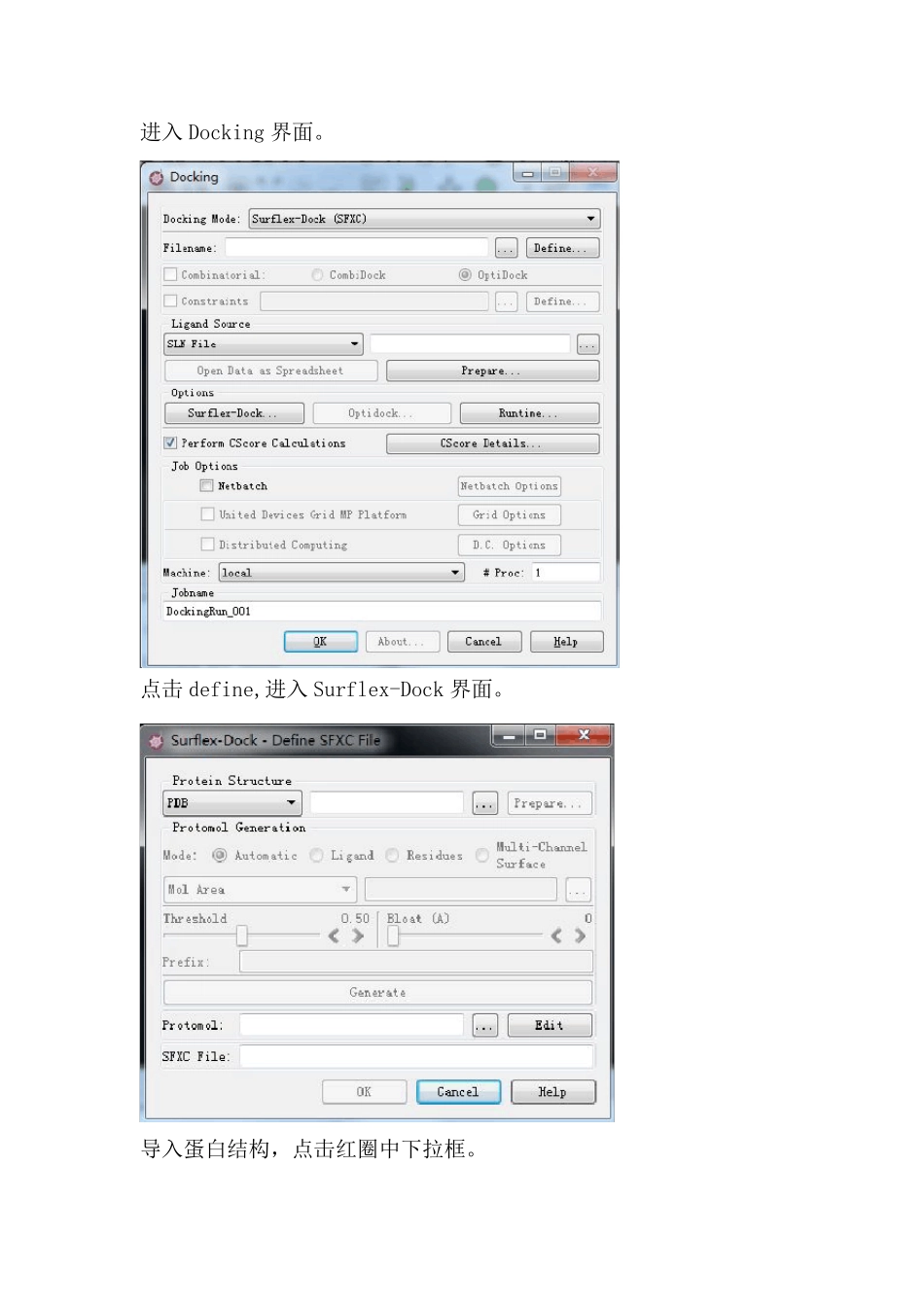
Sybyl 对接教程1.先建立存放需要对接的蛋白和小分子的文件夹。我建立的docking_sybyl 文件夹。文件夹里面放有靶点蛋白和需要对接的小分子,如下图。设置对接文件夹路径:Options→set→Default Directory找到docking_sybyl 文件夹,点击OK。下面开始对接:Application→ Docking Suite→ Docking Ligands进入Docking 界面。点击 define,进入Surflex-Dock 界面。导入蛋白结构,点击红圈中下拉框。进入docking_sybyl 文件夹。选择 3dlg.pdb 点击 OK。这样蛋白已经导入sybyl 窗口。接下来对蛋白。点击 Prepare。点击Extract Ligand Substructures(提取小配体)。选择小配体GWE999,点击OK。点击Remove Substructures(删去无关的水分子和离子)点击OK到此蛋白已经准备好。点击Return。点击OK。点击Generate,产生活性位点。即产生protomol,3dlg_H-L-0.50-0-protomol.mol2点击OK。在Ligand source 下拉框中有常见的小分子格式可以选择,看你要对接的小分子是什么格式就选什么格式。我的是mol2 格式就选择mol2 file点击红圈中下拉框,选择需要对接的小分子。选择AMD.mol2 点击OK。Proc 根据你的电脑的核数来确定,我的是两核,填2。四核填4。Jobname 可以根据习惯自己命名。点击 OK,开始对接。等待。。。对接完后出现Result Browser 界面。View 选择DockingRun1_site.mol2点击小分子AMD,出现结合模式。点击save result设置如下:每个配体保留3 个构象点击OK。显示小分子前3 个构象选择第一行,显示蓝色,点击3D。点击选择M2:3dlg,点击Specify。点击设置显示的颜色风格等。还可以保存纯蛋白和对接后的小分子,用来做分子动力学。点击AMD点击第二个点击Save这里说明一下:可以直接把删去小配体,水分子和离子的纯蛋白拿来做分子动力学。因为在对接过程中蛋白的构象并不变,所谓的半柔性对接。只有小分子的构象在变。