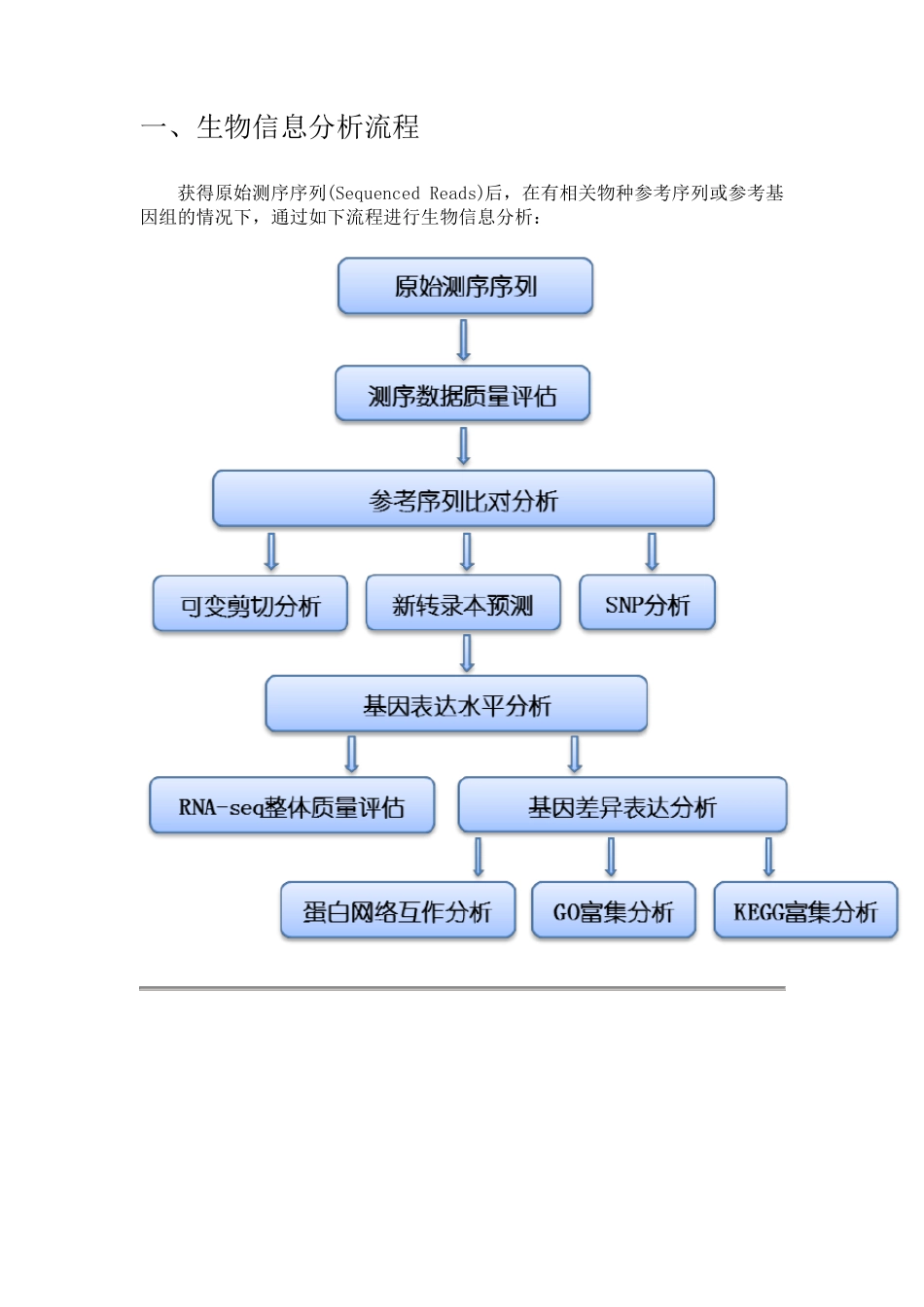
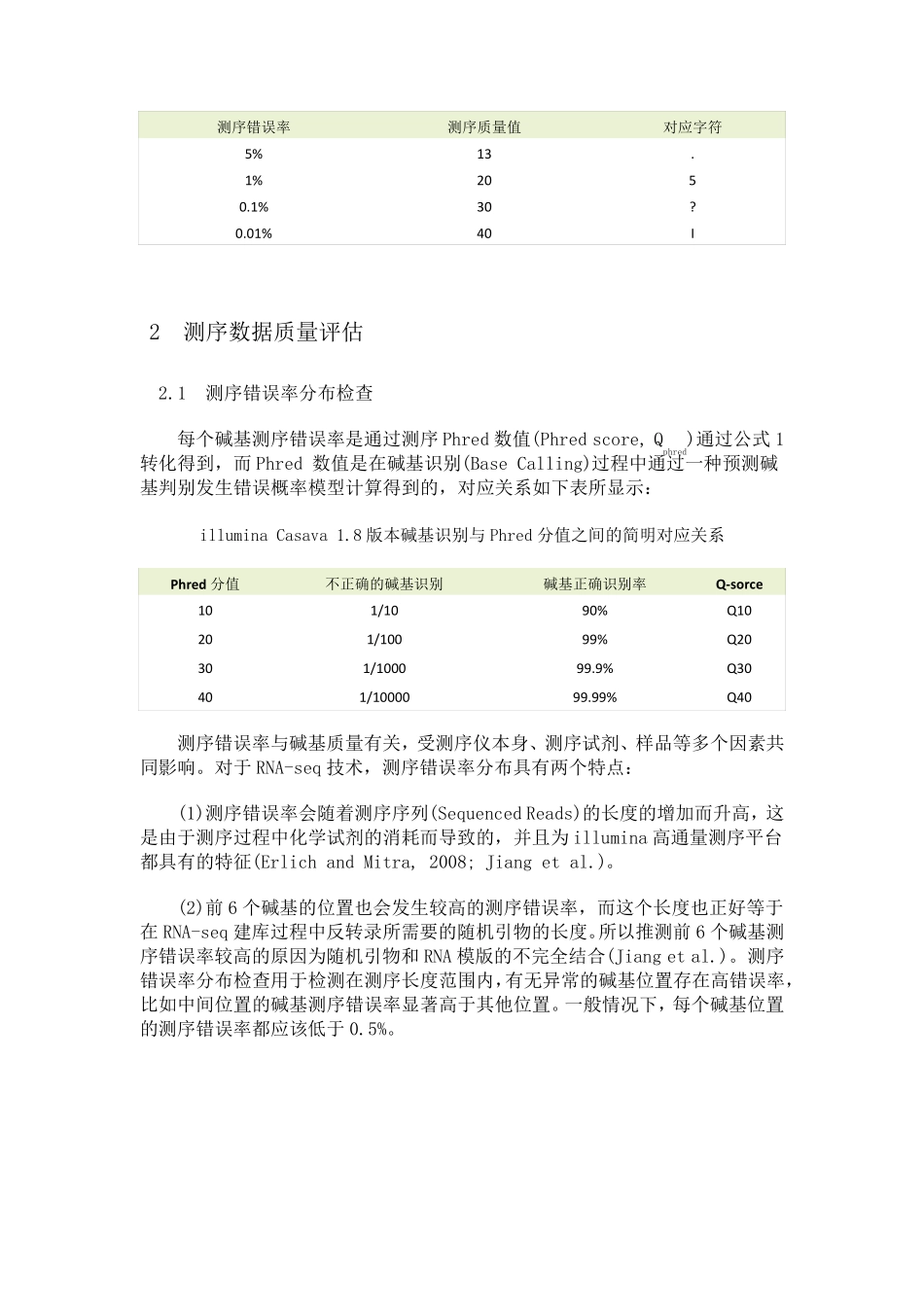
一、生物信息分析流程 获得原始测序序列(Sequenced Reads)后,在有相关物种参考序列或参考基因组的情况下,通过如下流程进行生物信息分析: 二、项目结果说明 1 原始序列数据 高通量测序(如illumina HiSeqTM2000/MiSeq 等测序平台)测序得到的原始图像数据文件经碱基识别(Base Calling)分析转化为原始测序序列(Sequenced Reads),我们称之为Raw Data 或Raw Reads,结果以FASTQ(简称为fq)文件格式存储,其中包含测序序列(reads)的序列信息以及其对应的测序质量信息。 FASTQ 格式文件中每个read 由四行描述,如下: @EAS139:136:FC706VJ:2:2104:15343:197393 1:Y:18:ATCACG GCTCTTTGCCCTTCTCGTCGAAAATTGTCTCCTCATTCGAAACTTCTCTGT + @@CFFFDEHHHHFIJJJ@FHGIIIEHIIJBHHHIJJEGIIJJIGHIGHCCF 其中第一行以“@”开头,随后为illumina 测序标识符 (Sequence Identifiers) 和描述文字 (选择性部分);第二行是碱基序列;第三行以“+”开头,随后为illumina 测序标识符(选择性部分);第四行是对应序列的测序质量(Cock et al.)。 illumina 测序标识符详细信息如下: EAS139 Unique instrument name 136 Run ID FC706VJ Flowcell ID 2 Flowcell lane 2104 Tile number within the flowcell lane 15343 'x'-coordinate of the cluster within the tile 197393 'y'-coordinate of the cluster within the tile 1 Member of a pair, 1 or 2 (paired-end or mate-pair reads only) Y Y if the read fails filter (read is bad), N otherwise 18 0 when none of the control bits are on, otherwise it is an even number ATCACG Index sequence 第四行中每个字符对应的ASCII 值减去 33,即为对应第二行碱基的测序质量值。如果测序错误率用 e 表示,illumina HiSeqTM2000/MiSeq 的碱基质量值用Qphred表示,则有下列关系: 公式一: Qphred = -10log10(e) illumina Casava 1.8 版本测序错误率与测序质量值简明对应关系如下: 测序错误率 测序质量值 对应字符 5% 13 . 1% 20 5 0.1% 30 ? 0.01% 40 I 2 测序数据质量评估 2.1 测序错误率分布检查 每个碱基测序错误率是通过测序Phred 数值(Phred score, Qphred)通过公式1转化得到,而Phred 数值是在碱基识别(Base Calling)过程中通过一种预测碱基判别发生错误...