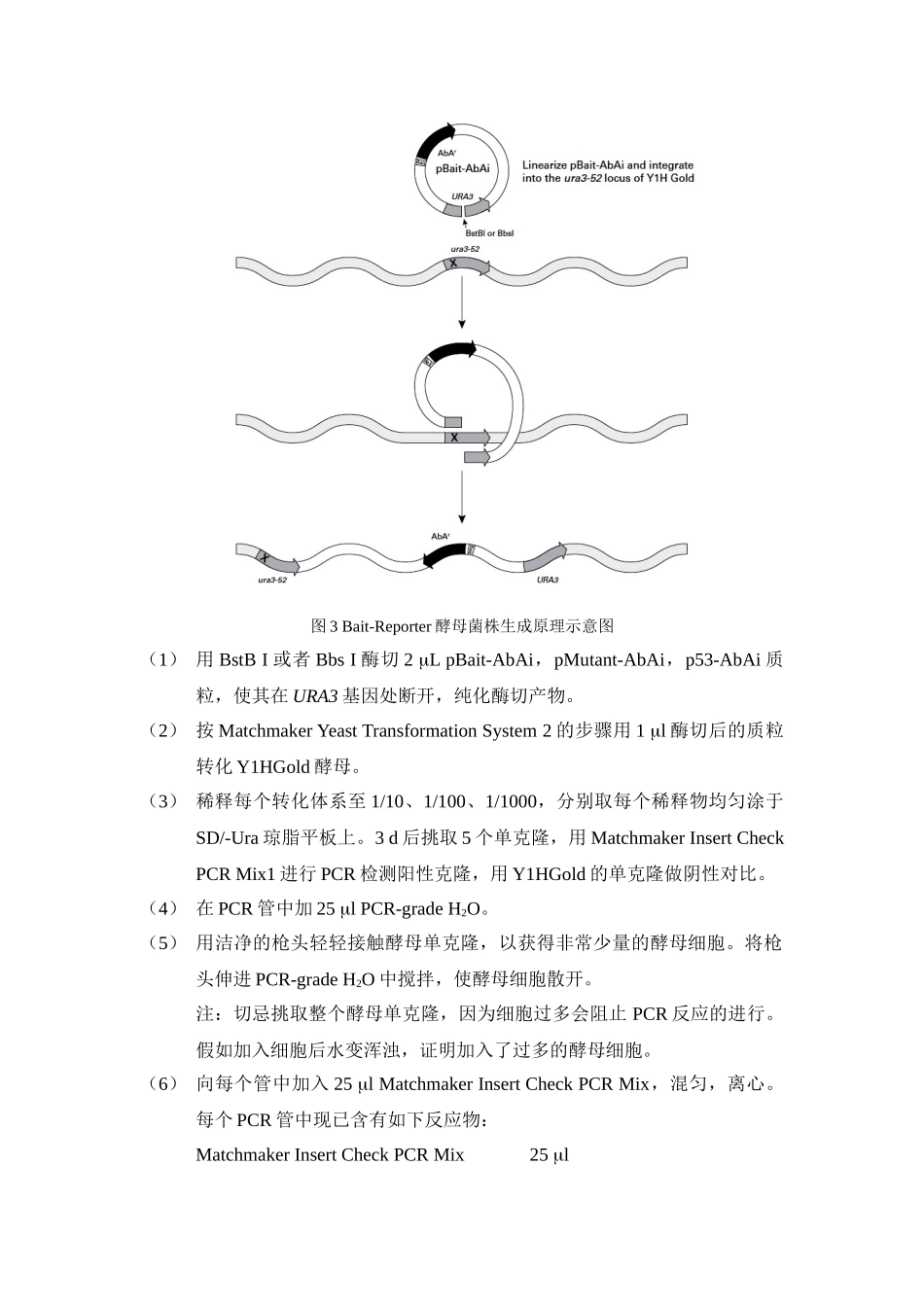
1 pBait-AbAi 载体的构建(酵母报道子的构建)注:酵母报道子(pBait-AbAi)包含目的顺式作用元件的一个或多个拷贝,且插入到 pAbAi 载体 AbAir报告基因的上游。大量讨论表明最有效的构建应包含目的 DNA 三个以上的首尾连接的拷贝。首尾连接的拷贝产生方式很多,但对于长度小于 20 bp 的调控元件,人工合成寡核苷酸是最方便可靠的途径。(1) 设计并合成包含目的序列的两条反向平行的寡核苷酸序列,且两端加上与 pAbAi 载体酶切产物一致的粘性末端(建议合成一个目的序列的突变序列作为对比,以排除可能的假阳性)。(2) 用 TE buffer 溶解寡核苷酸至终浓度 100 mol/L。(3) 将正向链和反向链根据 1:1 的比例混合(退火后的双链寡核苷酸最大浓度为 50 mol/L)。(4) 95 C 保温 30 s,去除二级结构。(5) 72 C 保温 2 min,37 C 保温 2 min,25 C 保温 2min。注:缓慢退火,有助于双链寡核苷酸的形成。(6) 冰上放置。退火后的产物可贮存在-20 C 冰箱备用。(7) 酶切 1 L pAbAi 载体,用凝胶回收纯化或柱纯化的方式纯化酶切产物。注:回收前,可用琼脂糖凝胶检测是否酶切完全。(8) 将退火后的寡核苷酸稀释 100 倍至终浓度为 0.5 mol/L。(9) 在连接反应管中加入如下成分:pAbAi 载体(50 ng/L) 1.0 Lannealed oligonucleotide (0.5 mol/L) 1.0 L10×T4 DNA ligase buffer 1.5 LBSA(10 mg/mL) 0.5 LNuclease-free H2O 10.5 LT4 DNA ligase (400 U/L) 0.5 L总体积 15 L 注:假如有必要,可用 1 L nuclease-free H2O 代替寡核苷酸作为阴性对比。(10) 将反应体系室温放置连接 3 h,转化 E coli,采纳常规方法检测阳性克隆。注:可用酶切或测序进行检测。2 质粒转化酵母细胞,生成 Bait-Reporter 酵母菌株(图)图 3 Bait-Reporter 酵母菌株生成原理示意图(1) 用 BstB I 或者 Bbs I 酶切 2 L pBait-AbAi,pMutant-AbAi,p53-AbAi 质粒,使其在 URA3 基因处断开,纯化酶切产物。(2) 按 Matchmaker Yeast Transformation System 2 的步骤用 1 l 酶切后的质粒转化 Y1HGold 酵母。(3) 稀释每个转化体系至 1/10、1/100、1/1000,分别取每个稀释物均匀涂于SD/-Ura 琼脂平板上。3 d 后挑取 5 个单克隆,用 Matchmaker Insert Check PCR Mix1...