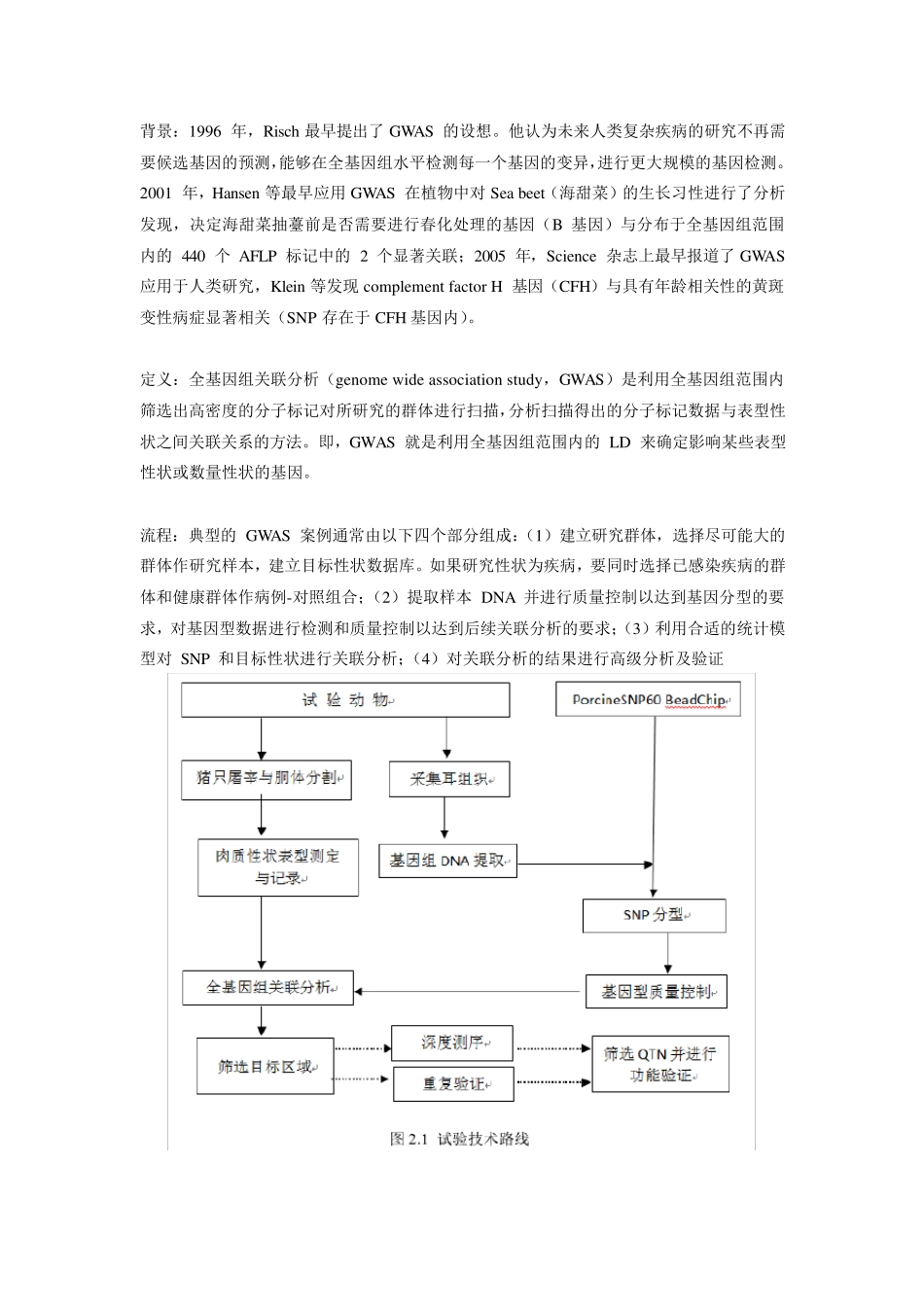
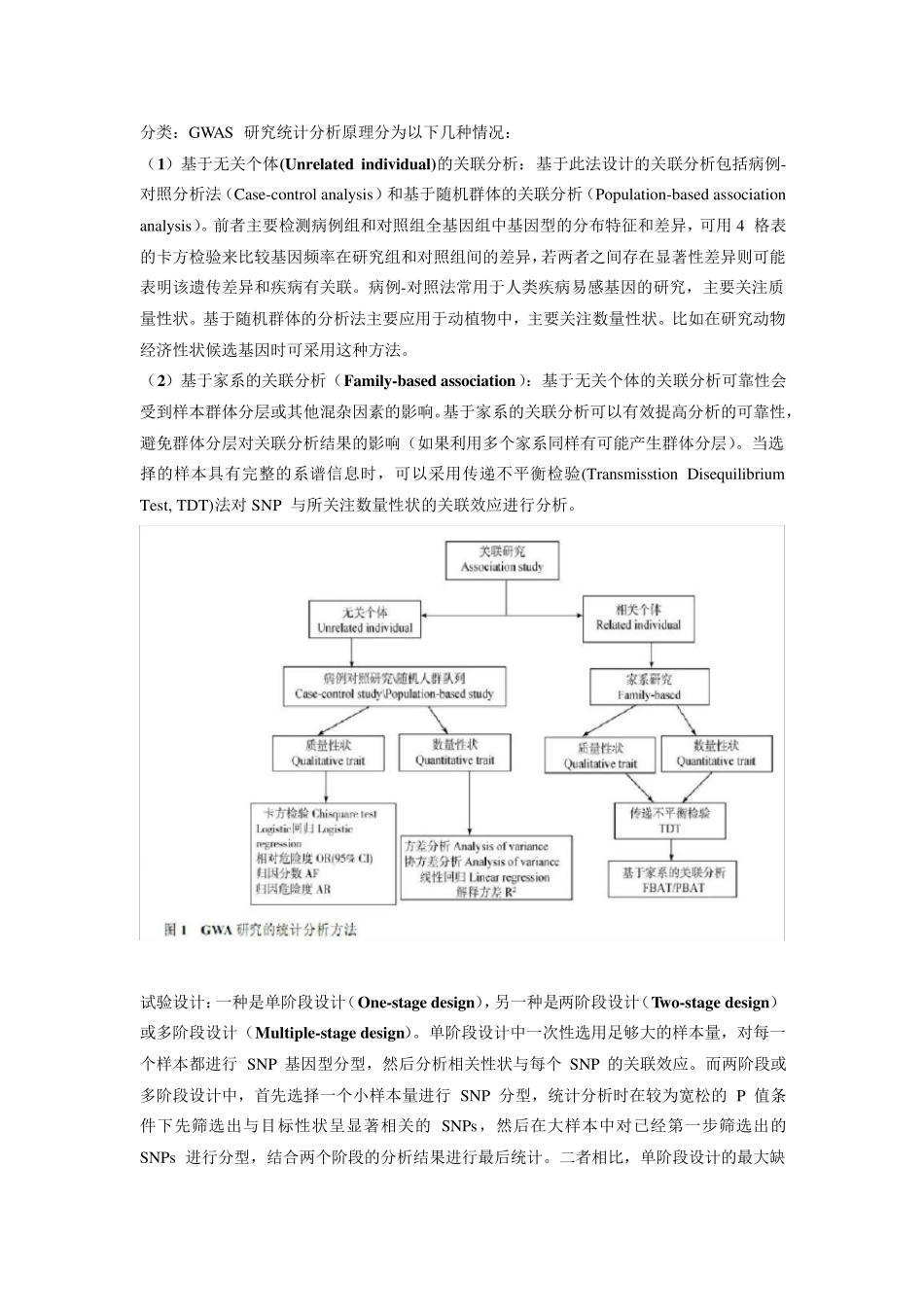
背景:1996 年,Risch 最早提出了GWAS 的设想。他认为未来人类复杂疾病的研究不再需要候选基因的预测,能够在全基因组水平检测每一个基因的变异,进行更大规模的基因检测。2001 年,Hansen 等最早应用 GWAS 在植物中对 Sea beet(海甜菜)的生长习性进行了分析发现,决定海甜菜抽薹前是否需要进行春化处理的基因(B 基因)与分布于全基因组范围内的 440 个 AFLP 标记中的 2 个显著关联;2005 年,Science 杂志上最早报道了GWAS 应用于人类研究,Klein 等发现 complement factor H 基因(CFH)与具有年龄相关性的黄斑变性病症显著相关(SNP 存在于 CFH 基因内)。 定义:全基因组关联分析(genome wide association study,GWAS)是利用全基因组范围内筛选出高密度的分子标记对所研究的群体进行扫描,分析扫描得出的分子标记数据与表型性状之间关联关系的方法。即,GWAS 就是利用全基因组范围内的 LD 来确定影响某些表型性状或数量性状的基因。 流程:典型的 GWAS 案例通常由以下四个部分组成:(1)建立研究群体,选择尽可能大的群体作研究样本,建立目标性状数据库。如果研究性状为疾病,要同时选择已感染疾病的群体和健康群体作病例-对照组合;(2)提取样本 DNA 并进行质量控制以达到基因分型的要求,对基因型数据进行检测和质量控制以达到后续关联分析的要求;(3)利用合适的统计模型对 SNP 和目标性状进行关联分析;(4)对关联分析的结果进行高级分析及验证 分类:GWAS 研究统计分析原理分为以下几种情况: (1)基于无关个体(Unrelated individual)的关联分析:基于此法设计的关联分析包括病例-对照分析法(Case-control analy sis)和基于随机群体的关联分析(Popu lation-based association analy sis)。前者主要检测病例组和对照组全基因组中基因型的分布特征和差异,可用 4 格表的卡方检验来比较基因频率在研究组和对照组间的差异,若两者之间存在显著性差异则可能表明该遗传差异和疾病有关联。病例-对照法常用于人类疾病易感基因的研究,主要关注质量性状。基于随机群体的分析法主要应用于动植物中,主要关注数量性状。比如在研究动物经济性状候选基因时可采用这种方法。 (2)基于家系的关联分析(Family-based association):基于无关个体的关联分析可靠性会受到样本群体分层或其他混杂因素的影响。基于家系的关联分析可以有效提高分析的可靠性,避免群体分层对关联分析...