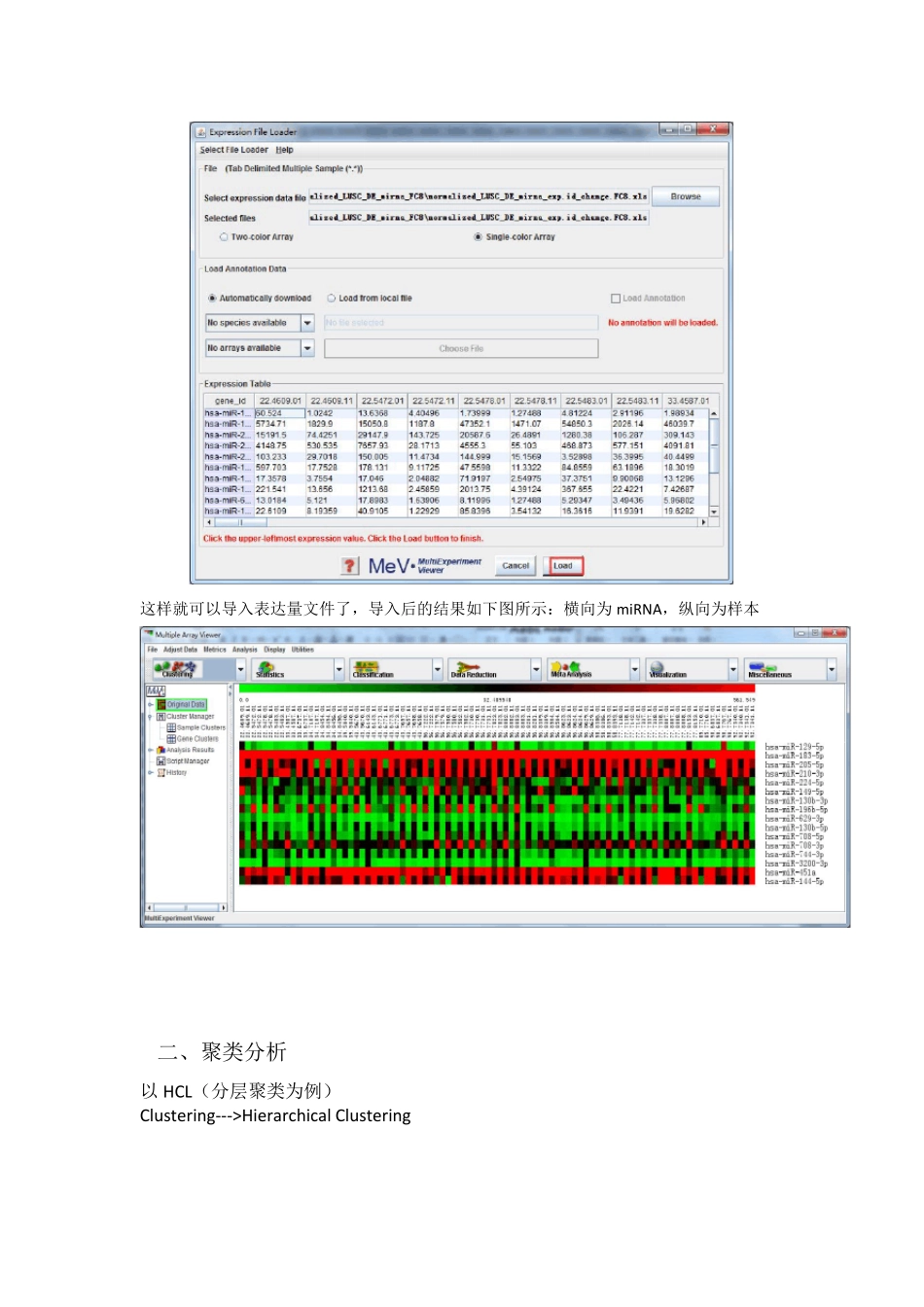
MeV 4.9.0 手册-热图绘制主要是进行热图的绘制、基本调整、高级调整以及小部分拓展。一、导入数据如:导入 mirna 在样本中的表达量数据File--->Load Data--->Brow se(选择输入文件)--->Load这样就可以导入表达量文件了,导入后的结果如下图所示:横向为miRNA,纵向为样本二、聚类分析以HCL(分层聚类为例)Clu stering--->Hierarchical Clu stering选择分层聚类后,在弹出来的对话框(如下图)里,可以选择是否基因聚类、是否样本聚类以及聚类时的度量,默认为 Pearson Correlation。我们这里基因与样本均聚类。点击 OK 后,就会发现左边指示栏里多出HCL(1)双击HCL(1)图标,可展开该目录,点击HCL Tree 可查看聚类结果。如下图所示:三、聚类结果调整由上图发现结果不是很好看,需要对结果对图形进行调整:⑴ 数据的调整(Adjust Data)可以对表达量数据横向标准化(Row )、纵向标准化(Columns)或者取 log 值。我们一般选择横向标准化,选择完这个标准后,结果如下:⑵ 结果的展示(Display)发现调整完数据后,结果不是很好看,我们需要将展示的结果进行调整。主要包括可热图颜色的调整(Color Scheme)、标尺范围的改变(Set Color Scale Limits)、标签字体大小(Set Font Size)、标签字体样式(Set Font Type)以及热图里每一个小方格的尺寸(Set Element Size)。比如:标尺范围的调整在弹出来的对话框中,可将方框里面的数值调整成与前面指示的数值相同:点击确认后,结果如下:可见,这时结果已经比较好了,再调整字体大小样式即可。四、高级调整⑴ 对聚类结果主要是样本树、基因树的细微调整:在图形显示区右键,可选择 GeneTree properties 或 SampleTree properties随便选择一个properties,假设选择SampleTree properties,弹出如下所示对话框,可选择样本树的高度、分支,节点高度标尺是否显示等。我们可以将两个对勾均去掉。结果如下图所示,可以发现显示树高度的标尺没有了,图会显得更美观。修改前修改后⑵ 给样本或基因添加标尺有时样本量太大,我们无法一个一个看样本名称看是什么样本,这时我们可以将同一类型的样本添加上同一标尺。同理,同种类型的基因也可以标识同样的标尺。例:给样本添加标尺选中同一类型的样本(被选中的树杈会变成红色),之后右键,选择 Store Clu ster。选择Store Clu ster 后会弹出如下对话框:我们可将这个Clu ster 命名为Control_Sample,颜色选为蓝色。...