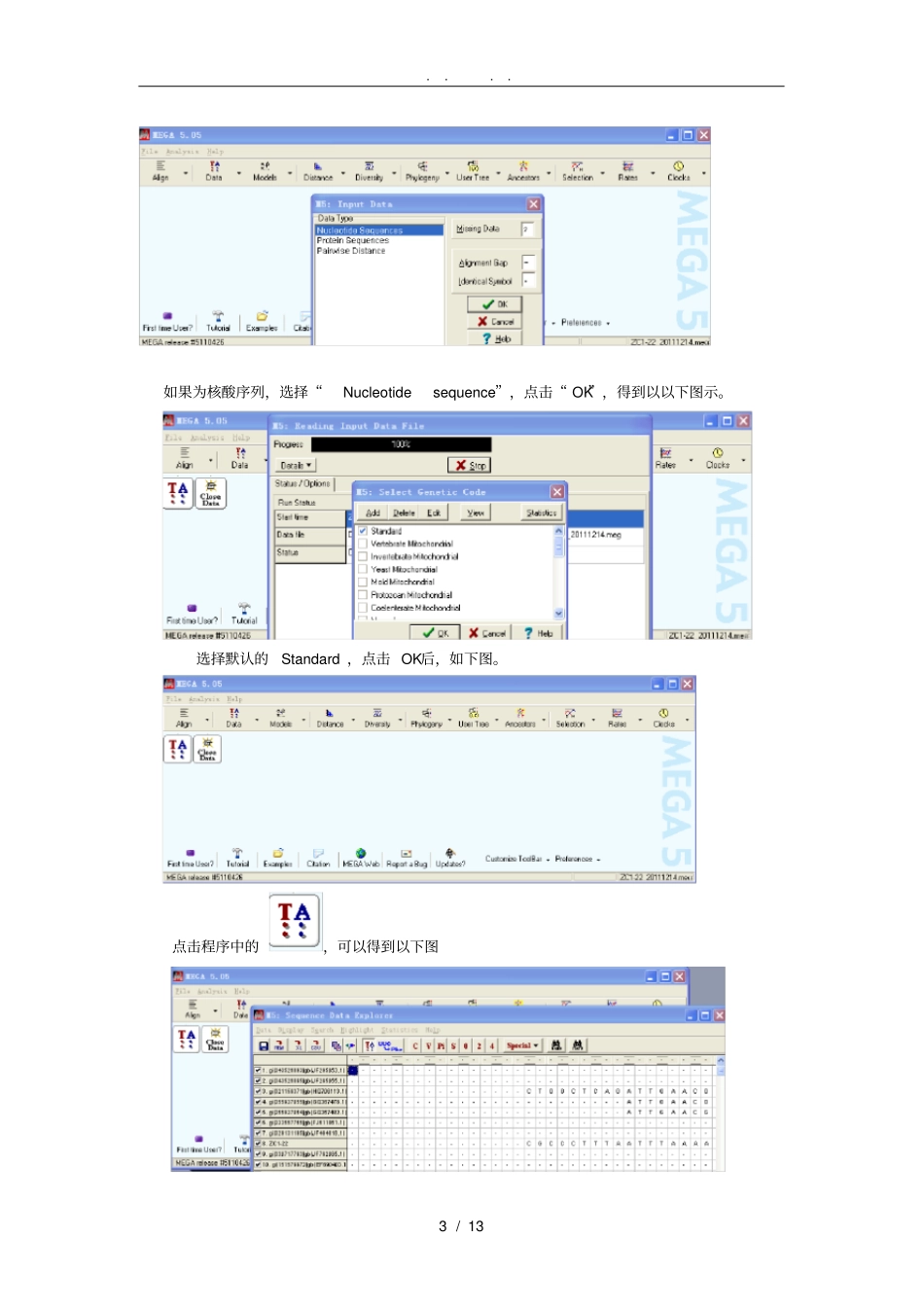
. . . . 1 / 13 如何用 MEGA和 Clustalx构建进化树MEGA是一个关于序列分析以与比较统计的工具包,从3.1 版本到后来的4.0 版本一直都广为大家熟悉, 现在推出了Mega5.0 版本。功能比以前多有改进。 现主要介绍使用Mega 5.0构建系统进化树的方法。供大家参考。用 MEGA构建进化树有以下步骤:1、测序:将克隆扩增测序得到的16S rDNA 序列进行测序。2、NCBI 上做 Blast .ncbi.nlm.nih.gov/blast/Blast.cgi找到相似度最高的几个序列,确定一下你分离的细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定你分离得到的就是Blast到的那个,然后寻找相似性最高的细菌,通常把该属的序列 (Fasta 格式文件)下载下来, 或点击 GenBank登录号,复制 FSATA格式,整合在一个*.txt文档中(单独建立一个文件夹存放,后面的很多文件会自动装入该文件夹),如>#### AGGCTTAACACATGCAAGTCGAGCGGAGCGAGGGTGCTTGCACCTTAGCTTAGCGGCGGACGGGTGAGTAATGCTTAGGAATCTGCCTATTAGTGGGGGACAACATTCCGAAAGGAATGCTAATACCGCATACGCCCTACGGGGGAAAGCAGGGGATCTTCGGACCTTGCGCTAATAGATGAGCCTAAGTCGGATTAGCTAGTTGGTGGG >gi|289469964|gb|GU388381.1| Acinetobacter tandoii strain DSM 14970 16S ribosomal RNA gene, partial sequence ACTTAGCGGCGGACGGGTGAGTAATGCTTAGGAATCTGCCTATTAGTGGGGGACAACATTCCGAAAGGGATGCTAATACCGCATACGCCCTACGGGGGAAAGCAGGGGATCTTCGGACCTTGCGCTAATAGATGAGCCTAAGTCGGATTAGCTAGTTGGTGGGGTAAAGGCCTACCAAGGCGACGATCTGTAGCGGGTCTGAGAGGATGA⋯⋯⋯⋯⋯⋯⋯⋯⋯⋯. 参考序列选择须知:1、不选非培养 (unclutured)微生物为参比;2、不选未定分类地位的微生物,最相近的仅作参考;c,在保证同属的前提下,优先选择 16S rDNA全长测序或全基因组测序的种;d,每个种属选择一个参考序列,如果自己的序列中同一属的较多,可适当选择两个参考序列。3、使用 clustalx1.83进行序列比对打开压缩文件clustalx1.83,运行其中的clustalx.exe文件,如图:点击 File/load sequences,将整理好的 *.txt序列文件导入clustalx1.83,如图. . . . 2 / 13 接着点击 Alignment/Do Complete Aligment 程序自动运行, 得出结果, 自动输出 *.aln和* .dnd 为后缀的两个文件, 并自动存入你 *.txt文件所在的文件夹。序列比对也可以直接用MEGA来做。4、运行程序MEGA 5.0,如以下图所示:点击:File导入 Clustal程...