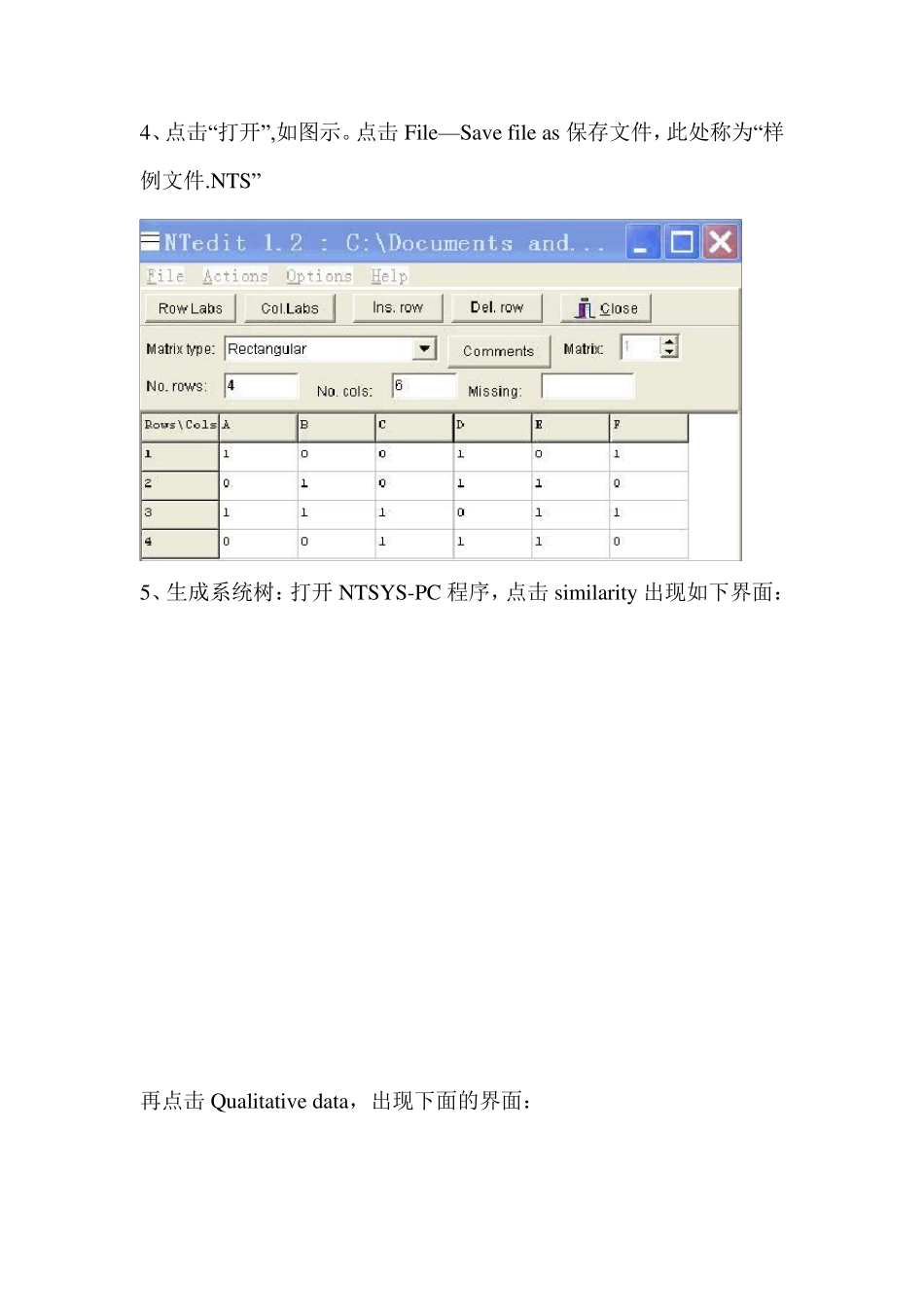
NTSYS-PC 使用说明 1.生成矩阵:在ex cel 按如下规则输入数据,A1=1 表示数据类型为矩阵,B1=4 表示扩增的总条带数(行数),C1=6 表示样本数(列数),D1=0 表示无缺失数据。第二行表示的是样本名称。从第三行开始的A 列表示引物名称。见下图: 输入完毕后,将文件以Microsoft ex cel 5.0/95 工作薄的格式存盘。此处称为“样例文件.x ls” 2、打开 出现如下界面: 3、点击 File— Open file in grid,如图示: 4、点击“打开” ,如图示。点击 File— Sav e file as 保存文件,此处称为“样例文件.NTS” 5、生成系统树:打开 NTSYS-PC 程序,点击 similarity 出现如下界面: 再点击 Qu alitativ e data,出现下面的界面: 点击inpu t file,打开生成的样例文件.NTS,点击ou t file 起一个文件名(假设叫A),然后点击compu te 按钮。继续点击clu stering,出现下面的界面, 再点SHAN,出现如下界面: 点击inpu t file,打开文件A,点击ou t file 起一个文件名 B,将 in case of ties 中的 WARN 改为 FIND 然后点击compu te 按钮.出现如下界面: 点击上图中右下角的图标,即可生成聚类图如下: 图中的Matrix 数值可调,具体作用不清楚。系统默认样本数为30,若样本数量较多,可点击options—plot options 出现如下界面: 此处可调节样本数量、字体大小、图形中线条的粗细和颜色及标题。 6、生成 meg 文件:数据处理过程中产生的文件 B 是遗传相似系数 I,根据 D=1-I 在 ex cel 中计算得遗传距离 D,如图示: 将遗传距离 D 由“遗传距离.x ls”另存为“遗传距离.tx t”文件。将遗传距离.txt”修改为如图示的文件。再将其另存为“遗传距离.meg”格式文件。 7、生成聚类图:双击文件“遗传距离.meg”,出现如下界面: 点击Phylogeny—Construct Phylogeny—Neighor-Joining(NJ) [此处根据需要选择NJ、ME、UPGMA 任一种聚类方法],出现如下界面: 如上图示,点击Compu te 即可做出聚类图,如下图示: 点击view—Tree/Branch Style—Traditional/Radiation/Circle 选择所需聚类图形状。 在设置中可修改图中线条的粗细、颜色以及样本的代表形状、颜色等。