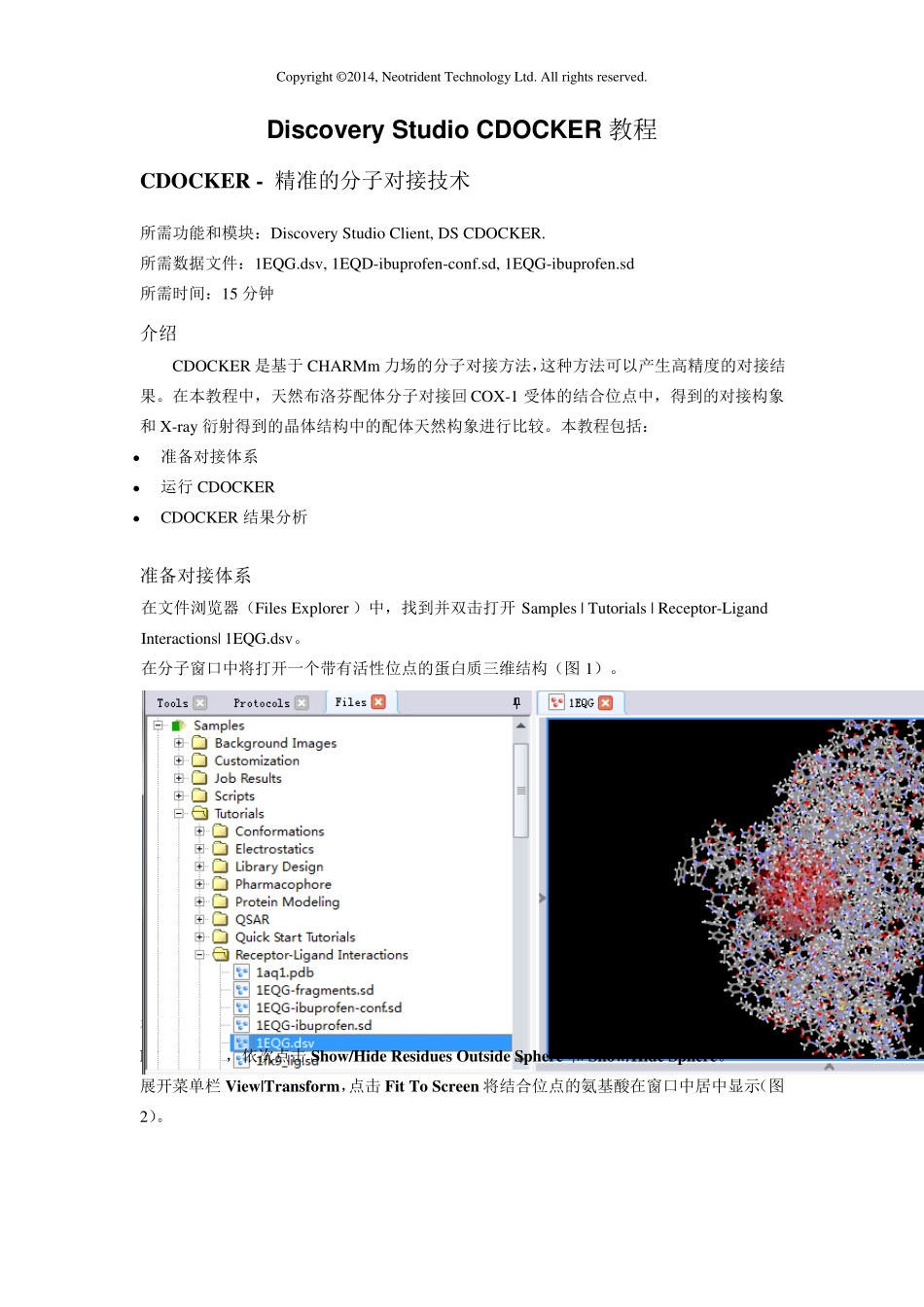
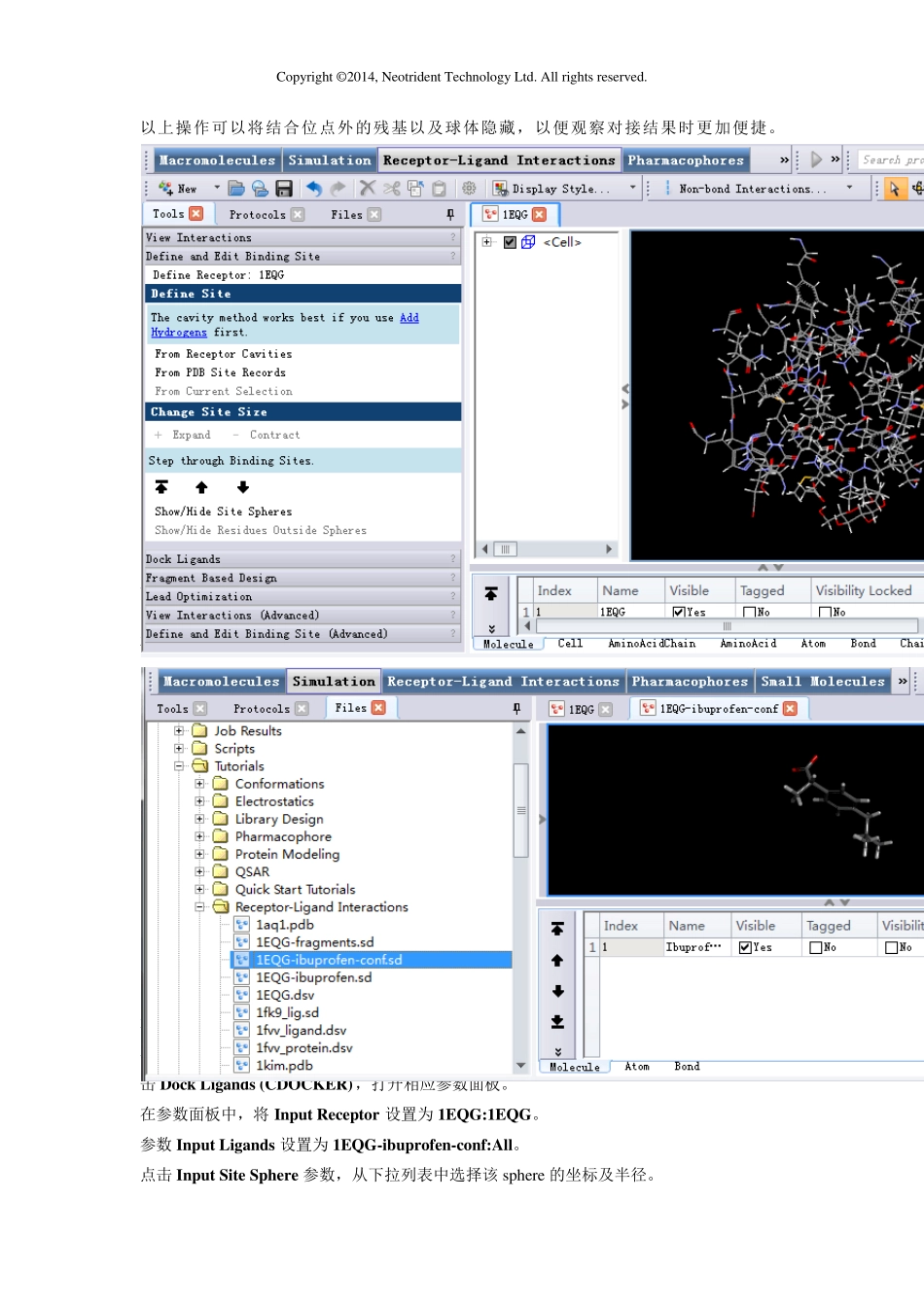
Copyright © 2014, Neotrident Technology Ltd. All rights reserved. DS CDOCKER Copyright © 2014, Neotrident Technology Ltd. All rights reserved Copyright © 2014, Neotrident Technology Ltd. All rights reserved. Discovery Studio CDOCKER 教程 CDOCKER - 精准的分子对接技术 所需功能和模块:Discovery Studio Client, DS CDOCKER. 所需数据文件:1EQG.dsv, 1EQD-ibuprofen-conf.sd, 1EQG-ibuprofen.sd 所需时间:15 分钟 介绍 CDOCKER 是基于CHARMm 力场的分子对接方法,这种方法可以产生高精度的对接结果。在本教程中,天然布洛芬配体分子对接回 COX-1 受体的结合位点中,得到的对接构象和X-ray 衍射得到的晶体结构中的配体天然构象进行比较。本教程包括: 准备对接体系 运行 CDOCKER CDOCKER 结果分析 准备对接体系 在文件浏览器(Files Explorer )中,找到并双击打开 Samples | Tutorials | Receptor-Ligand Interactions| 1EQG.dsv。 在分子窗口中将打开一个带有活性位点的蛋白质三维结构(图 1)。 图 1 蛋白质三维结构示意图 在工具浏览器(Tools Explorer )中,展开 Receptor-Ligand Interactions | Define and Edit Binding Site,依次点击 Show/Hide Residues Outside Sphere 和Show/Hide Sphere。 展开菜单栏 View|Transform,点击 Fit To Screen 将结合位点的氨基酸在窗口中居中显示(图2)。 Copyright © 2014, Neotrident Technology Ltd. All rights reserved. 以上操作可以将结合位点外的残基以及球体隐藏,以便观察对接结果时更加便捷。 图 2 蛋白质活性位点氨基酸 展开菜单栏 Files,点击 Open...,打开 Samples | Tutorials | Receptor-Ligand Interactions| 1EQG-ibuprofen-conf.sd文件。 将打开一个具有随机构象的布洛芬分子(图 3)。 图 3 配体小分子结构 运行 CDKCKER 在工具浏览器(Tools Explorer)中,展开 Receptor-Ligand Interactions | Dock Ligands,点击 Dock Ligands (CDOCKER),打开相应参数面板。 在参数面板中,将Input Receptor 设置为 1EQG:1EQG。 参数 Input Ligands 设置为 1EQG-ibuprofen-conf:All。 点击 Input Site Sphere 参数,从下拉列表中选择该 sphere 的坐标及半径。 Copyright © 2014, Neotri...