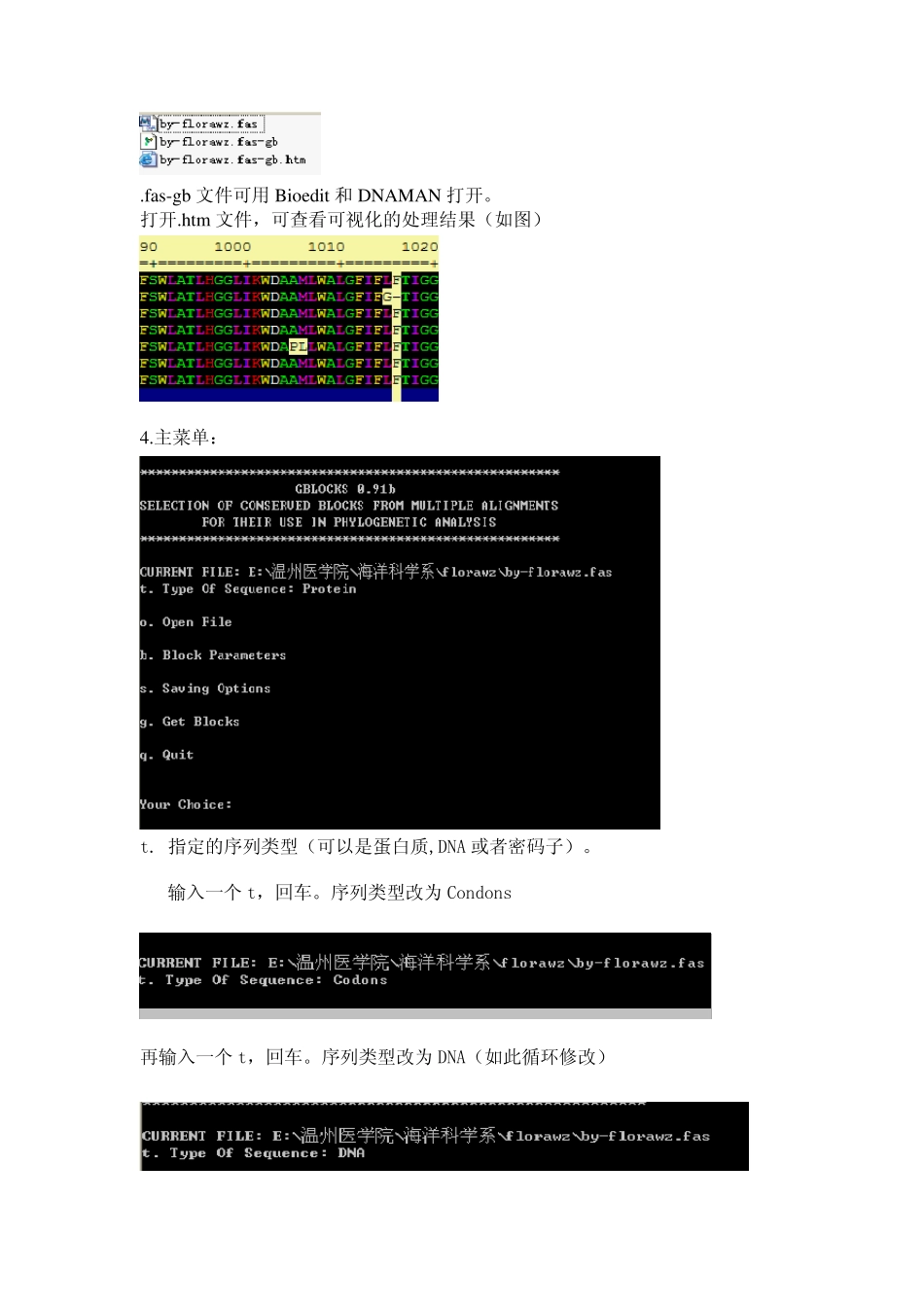
Gblocks 使用说明书(by florawz) 1.首先打开软件,进入主页面 2.输入O ,然后回车 ,对话框显示输入一个文件或路径 此时将比对好的(.fas)文件拖入对话框。对话框即出现该文件的路径(如图) 按回车,即导入该序列。对话框上部出现下列信息 3.快速比对:输入G,然后回车。在原比对文件所在文件夹内即可出现 Gblocks已经处理好的文件 .fas-gb 文件可用Bioedit和DNAMAN 打开。 打开.htm 文件,可查看可视化的处理结果(如图) 4.主菜单: t. 指定的序列类型(可以是蛋白质,DNA 或者密码子)。 输入一个 t,回车。序列类型改为 Condons 再输入一个 t,回车。序列类型改为 DNA(如此循环修改) o. 打开一个文件。必须为 NBRF/PIR 或 FASTA 格式 ,序列长度不限。打开 NBRF/PIR-格式的序列时,在序列备注第一行要注明序列类型 如: >P1;byflorawz ------MEYLLQEYLPILVFLGMASALAIVLILAAAVIAVRN--PDPEKVSAYECGFNAF D-DARMKFDVRFYLVSILFIIFDLEVAFLFPWAVSFASLS-DVAFWGLMVFLAVLTVGFA YEWKKGALEWA----------------------* (fas 格式则不需要,第一行直接为>byflorawz即可) 注意:在使用 Glocks 分析前,序列缺口必须先消除。 在将比对文件拖进改软件时,要去路径掉末尾的空格。 打开多个文件 :必须建立一个path 文件。输入各个相关文件的路径,在安装好的文件包内可以看到一个"paths"范例,用 word 打开此文件,即可看到各个文件的所在路径(如图) 多条比对序列的处理:如果所有的比对文件的路径都在一个paths 文件,且各个比对文件的序列条数,以及物种的顺序都是相同的,那么这些比对文件在最后的结果中可以连接起来。如果各个比对文件的序列条数不同,那么也可以一起处理,但是最后不能连接。 b. 显示 Block 限制性参数 (详情见下页). s. 显示保存菜单(详情见下页). g. 处理计算 q. 退出 5 .限定性参数菜单 1. 设置保守区域内的保守序列的至少数量, 必须大于序列个数一半,数值越大,所挑选的保守位点越少 7 个序列设置参数为 5 7 个序列设置参数为 4 7 个序列设置参数为 6 2. 设置保守区域两侧位置的保守序列的最少数量,必须大于或等于1 所设置的值,这个值越大,所选择的保守位点越少 7 个序列设置参数为6 7 个序列设置参数为5 3.保守区内一段非保守区的最大范围,数值越大,选择的保守位点越多。 设置为4 时(5-6-4-10) 设置为5 时(5-5-5-10) (5-5...