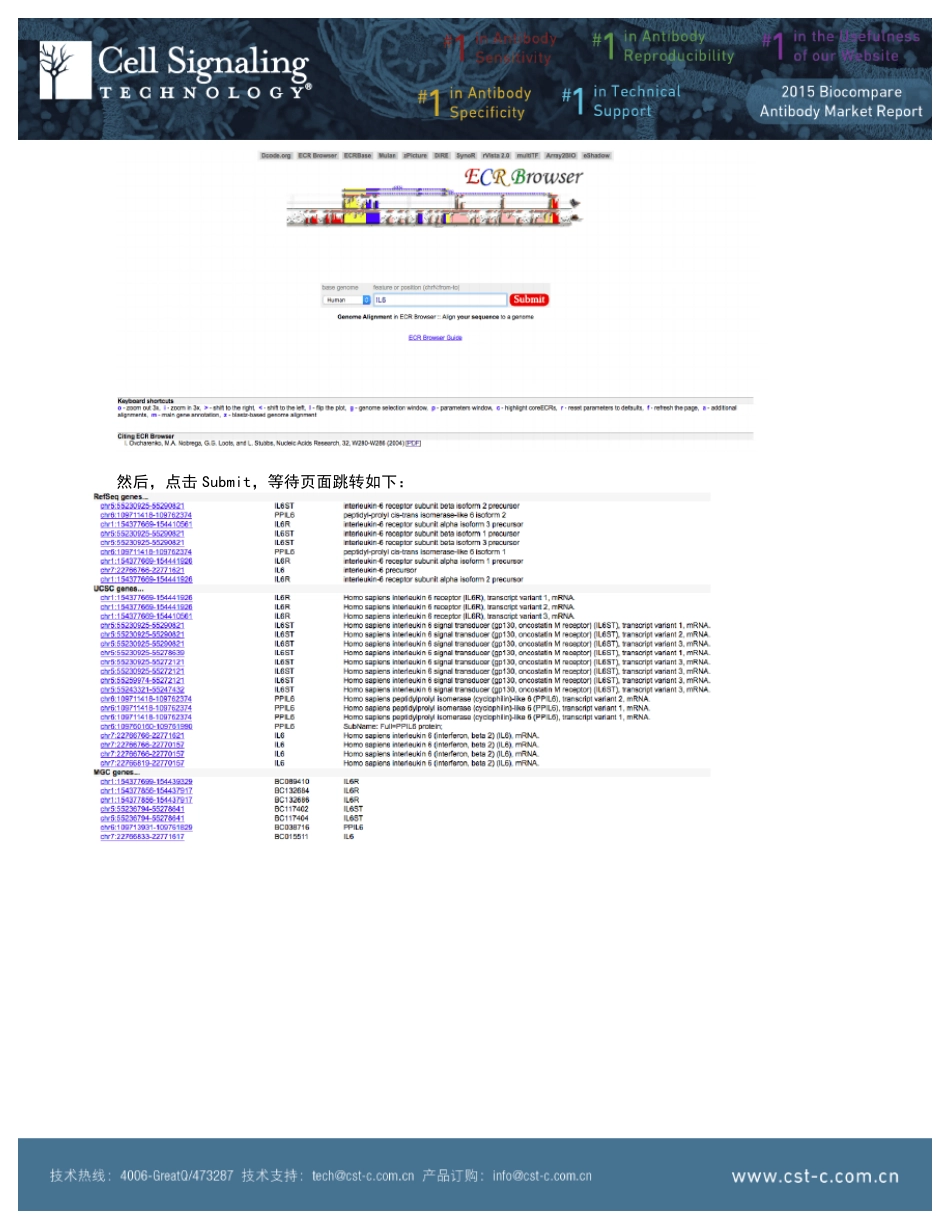
用ECRBrowser预测转录因子结合位点的方法 CST 中 国 公 司 学 术 经 理 李 振 亚 博 ⼠ 经常会有一些朋友因为转录相关研究而需要进行转录因子结合位点的预测,以通过染色质免疫沉淀(ChIP)方法或其他研究转录调控的方法进行验证。我在这里给大家分享一个我经常使用的在线工具—ECRBrowser(https://ecrbrowser.dcode.org),并介绍一下如何用这个工具进行转录因子结合位点的预测。 首先,打开网址,会跳转到一个界面,如下图所示: 然后,在搜索引擎选择你所要研究的物种,等待 3-5 秒钟,让页面更新: 然后,在第二个信息框内填写你想要预测转录因子结合位点的基因名称(如 IL6,可根据 NCBI 标准基因名称填入) 然后,点击 Submit,等待页面跳转如下: 在其中选择正确的对应基因名称,由于我一般喜欢用Refseq 数据库,所以我点击了套红的那个链接,即chr7:22766766-22771621。 等待页面跳转如下: 此时,这个页面显示的是整个IL6 基因的转录区域,即mRNA 全长(含内含子)所对应的基因组区域。注意,这个区域并不包括该基因的启动子区域。 那可能大家就要问了,我要预测转录因子结合位点,得需要基因启动子区域啊,那怎么做呢? 按照很多主流转录组学研究的核心启动子区纳入范围,都是在mRNA 的起始位点的上游 2kb 以内。我也是按照这个标准去获取核心启动子区域。对于这个基因IL6,如果要在IL6 假定的启动子区域搜索转录因子结合位点,那么需要手动更改基因组的起止位点。请大家注意,现在页面的显示区域是 chr7:22766766-22771621,起始位点是 22766766,终止位点是 22771621。由于这个基因位于正链(即从左向右转录),我按照上述核心启动子区域的选定标准,把基因组区域显示范围更改为:chr7:22764766-22766766。大家发现什么规律没有?对啦,就是原来基因组显示的起始位置对应的一串数字变成基因组显示区域终止位置的数字,然后将这串数字减去 2000,即得到需要的假定启动子区域起始位置对应的数字了! 那可能有人会问了,如果某一个基因位于负链呢?这个时候你首先会发现这个基因对应的mRNA 会显示为从右向左转录。然后再获取这样的基因假定的启动子区域时,就需要把原来基因组显示的终止位置对应的一串数字变成基因组显示区域起始位置的数字,然后将这串数字加上 2000,即得到需要的需要的假定启动子区域终止位置对应的数字了! 当然,大家可以选择更大的范围(如包含启动...