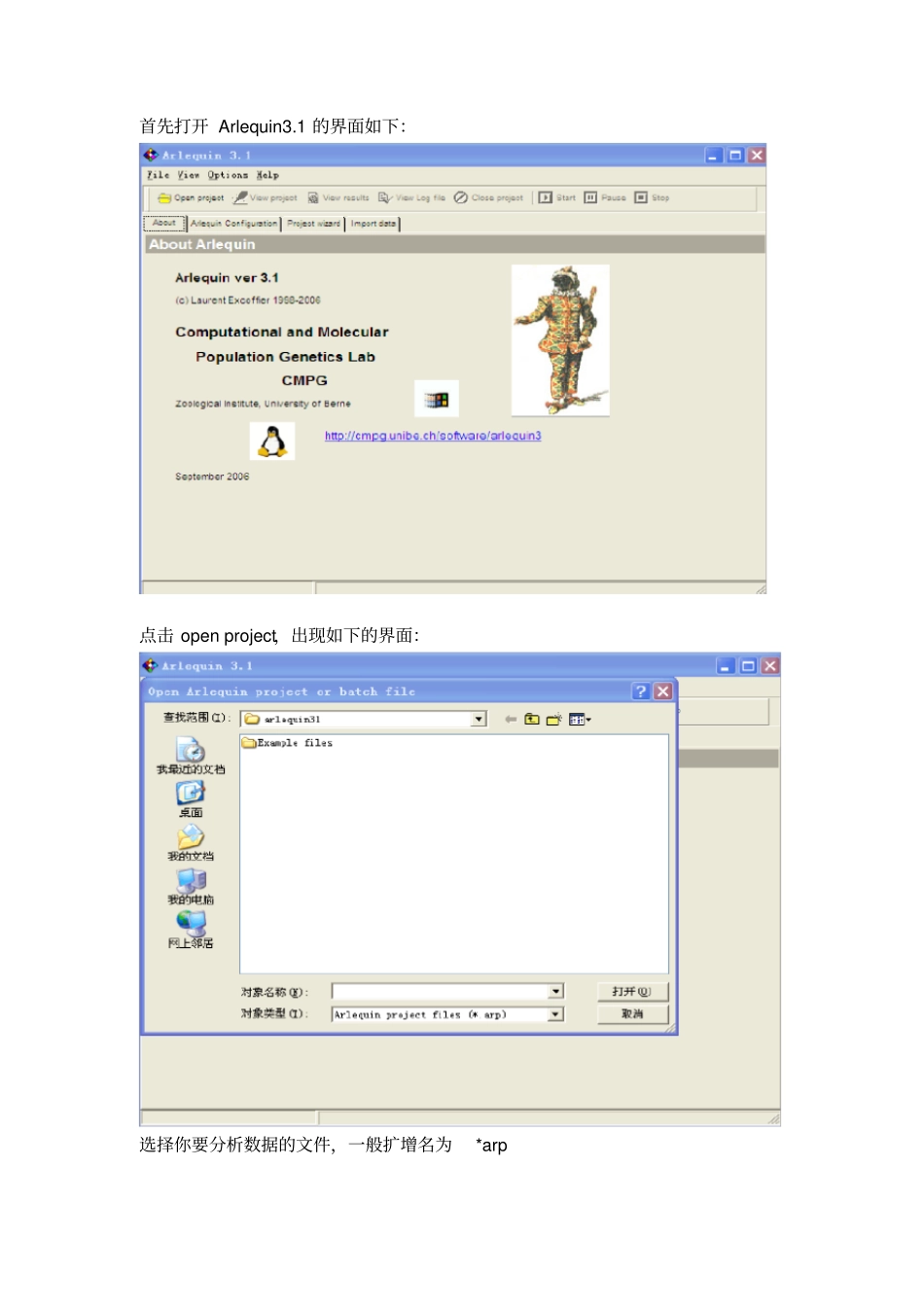
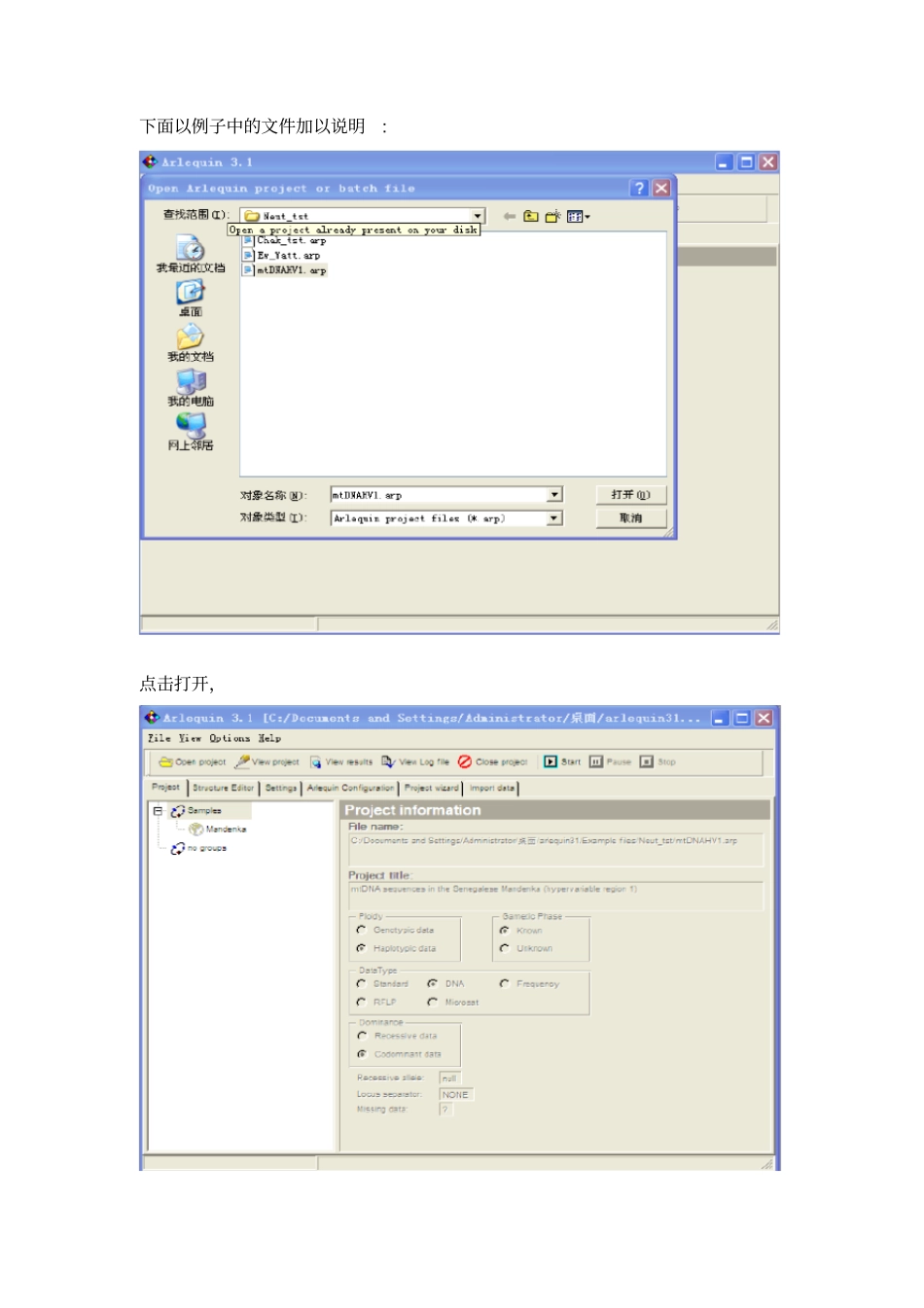
Arlequin3.1 的使用说明Arlequin 功能的概述Molecular diversity—分子多态性Mismatch distribution—错配分布Haplotype frequency estimation—单倍型频率估计Linkage disequilibrium —连锁不平衡: 检测不同位点上等位基因的非随机关联Hardy-Weinberg equilibrium—哈温—伯格平衡Tajima’s neutrality test— Tajima 中性检测Fu’ s neutrality test— Fu 中性检测Ewens-watterson neutrality test— Ewens-watterson中性检测以上三个中性检测都是基于无限位点模型,适用于DNA sequence和 RFLP 单倍型。Chakraboety’s amalgamation test— Chakraboety’s 融合检测,检测人群的均一性和同质性,和中性选择等。Minimu Spanning Network(MSN ,最小扩张树或称之为最小支撑树,给予分子差异。AMOV A—分子差异度分析,用以评测人群的遗传结构Parwise genetic distances—遗传距离的估计Exact test of population differentiation—检测随机交配群体单倍型的非随机分布Assignment test of genotype—通过估计等位基因平率将单个基因型分被盗特定的人群中。Arlequin3.1 分析的数据文件的格式必须是以*arq 为扩增名方法:用 Clustalx 比对后保存一个 PHY 格式的文件,在 DNAsp 中打开该文件,点击 Generate中的 Haplotype date file ,设置 considered,其余的参数不变, 在其中的 Generate中选其中的 Arlequin Haplotype List 即可保存下用 Arlequin3.1 分析的文件的格式 (在 import date中可以进行文件格式的转换,Arlequin 的数据转化为 Arlequin 、Genepop、Biosys、phylip、Mega、WinAmova)首先打开 Arlequin3.1 的界面如下:点击 open project,出现如下的界面:选择你要分析数据的文件,一般扩增名为*arp 下面以例子中的文件加以说明: 点击打开,Project title 所分析数据的名称Genotypic date 输入数据是单倍型还是双倍性Gametic phase:确定输入数据中配子片段是否已知Recessive date:确定输入数据是否为阴性Date type:输入数据的类型Missing date:缺失数据用什么字符表示对上面的例子进行数据设置点击 Setting 出现如下的界面点击 General setting情况如下:点击 Haplotype inference(单倍型频率),出现如下界面选中右侧的设置情况,如果输入的数据的形式属于配子片段已知的单倍型数据或基因型数据,则操作界面如下继续设置,点击Mole...