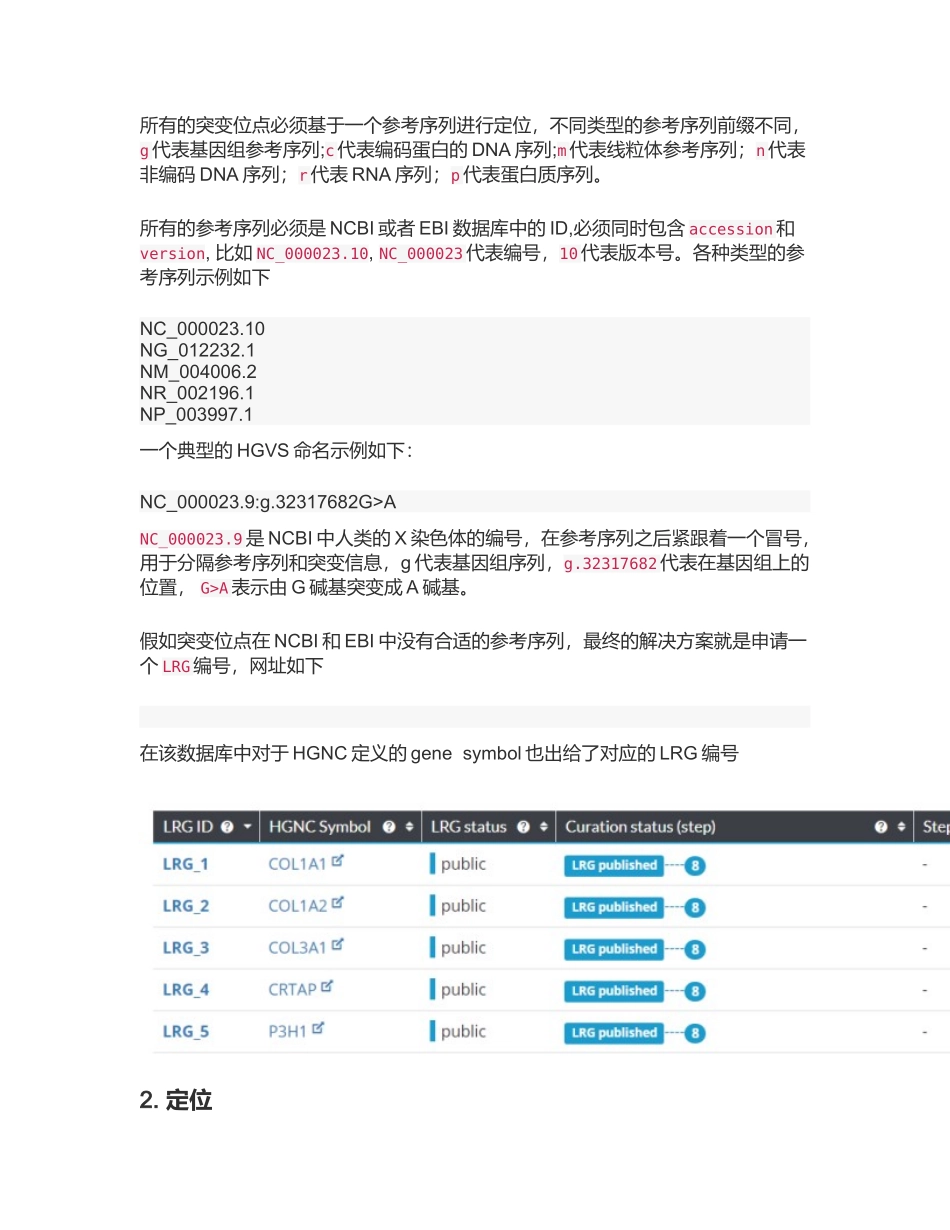
HGVS 制订的变异位点命名规则(source: Last time: 2025/12/24) 关注2025.05.26 14:26 字数 1834 阅读 1747 评论 0 喜爱 2 欣赏 1欢迎关注"生信修炼手册"!HGVS 指定了一套完整的变异位点命名规则,统一的命名方便了学术沟通与沟通。官网链接如下:对于所有的变异位点,划分成了 3 个层次1. DNA level2. RNA level3. Proteion level一个好的命名,至少要体现 2 个因素:变异位点的位置和造成的影响,HGVS 通个以下 3 个方面来定义一个变异位点1. reference sequence2. position3. variant type1. 参考序列所有的突变位点必须基于一个参考序列进行定位,不同类型的参考序列前缀不同,g 代表基因组参考序列;c 代表编码蛋白的 DNA 序列;m 代表线粒体参考序列;n 代表非编码 DNA 序列;r 代表 RNA 序列;p 代表蛋白质序列。所有的参考序列必须是 NCBI 或者 EBI 数据库中的 ID,必须同时包含 accession 和version, 比如 NC_000023.10, NC_000023 代表编号,10 代表版本号。各种类型的参考序列示例如下NC_000023.10NG_012232.1NM_004006.2NR_002196.1NP_003997.1一个典型的 HGVS 命名示例如下:NC_000023.9:g.32317682G>ANC_000023.9 是 NCBI 中人类的 X 染色体的编号,在参考序列之后紧跟着一个冒号,用于分隔参考序列和突变信息,g 代表基因组序列,g.32317682 代表在基因组上的位置, G>A 表示由 G 碱基突变成 A 碱基。假如突变位点在 NCBI 和 EBI 中没有合适的参考序列,最终的解决方案就是申请一个 LRG 编号,网址如下在该数据库中对于 HGNC 定义的 gene symbol 也出给了对应的 LRG 编号2. 定位对于突变位点而言,位置信息是基本信息之一。对于不同的参考序列,定位的策略也稍有差异。g 代表基因组,m 代表线粒体, p 代表蛋白质,这三种参考序列在定位时,都是从 1开始计数,写法为 g.1, m.1, p.1, 除此之外,不需要任何的修饰符号。c 代表编码蛋白的 DNA 序列,从起始密码子的第一个碱基开始计数,写法为 c.1, 只对 exon 区间进行计数,终点为终止密码子的最后一个碱基。对于起始密码子上游的碱基,采纳负号表示,比如 c.-1;对于终止密码子下游的碱基, 采纳*表示,比如 c.*1;在内含子区的变异位点要根据距离来决定,靠近内含子 5’末端的变异位点,要根据上游最近的外显子的最后一个碱基来定位,示例 c.87+4.上游最近的外显子...